768 种 MGISEQ 双端 Index 解决方案 (for MGI)
应用背景
随着二代测序通量的不断提升,Illumina 公司的 HiSeq 和 NovaSeq、以及华大智造的 MGISEQ/DNBSEQ 系列测序仪,均可一次产出数百 Gb 以上数据。为最大化利用测序通量,通行做法是多样本混合测序:即在文库构建环节为每个样本添加唯一标签序列(Index),并在测序后依据 Index 进行数据拆分。
在文库制备、测序和数据分析中的各个环节,包括合成错误或污染、操作不当、PCR 异常扩增、测序错误、生信分析错误等,均可能导致标签错配(Index Misassignment)。因此,研究人员应高度关注因数据串扰而导致的错误分析结论。例如,Illumina 测序平台的 ExAmp 扩增方式本身就会导致高达 0.25-7% 的标签错配率,但可使用双端唯一类型的接头(Unique Dual Index, UDI)极大缓解标签错配的影响[1]。
华大智造测序平台因使用 PCR-free 的 DNB 扩增方式,当使用单 Index 接头时的标签错配率较低(平均为 0.0004%,最高至 0.001%)。此时约 68% 的标签错配来自于测序错误,32% 来自于合成或操作的污染[2]。然而进行靶向捕获时,由于多个步骤均与标签错配的产生有关,因此串扰问题可能更为突出[3]。
纳昂达对含有不同 HPV 类型的宫颈癌细胞系分别使用 MGI 平台单端 Index 建库和 Illumina 平台双端 Index 建库,然后进行 5 – Plex 的靶向捕获测序,数据拆分结果如表 1 所示。与 Illumina 平台的 UDI 方案相比,单端 Index 拆分数据中存在跳跃污染。这也提示我们,在实际应用中,UDI 是应对标签错配污染最简单、可靠的方法。
表 1:细胞系经不同文库构建方式后的HPV检出分布

方案推荐
为优化解决标签错配问题,纳昂达科技已于 2020 年推出 MGI 平台的 96 种双端唯一标签接头模块:MDI(MGISEQ Unique Dual Index)系列产品。近期,该系列产品又进一步扩展至 768 种!MDI 的引入使得文库两端的 Index 序列均是唯一的,且一对一组合,不存在共用。只有两端均带有完全正确 Index 序列的 Reads 才能进入后续的样本分析,从而剔除标签错配的 Reads,有效避免样本之间的数据串扰。
独家专利设计!(专利号:CN111910258B)
双端唯一Index for MGI,多达768种
随着“全球日生产能力最强”的基因测序仪 DNBSEQ-T7 的陆续交付,单 lane 的数据产出已高达 1440 Gb。若以全外显子测序应用为例(如 Exome Plus Panel v2.0),达到约 250x 平均深度时所需数据量为 20 Gb,需要 72 种 Index;以泛实体瘤检测应用(如NanOnco Plus Panel v2.0)为例,达到约 1000x 平均深度时所需数据量为 5 Gb,则至少需要 288 种 Index。相对低通量的 Index 设计方案显然无法满足更多的混样需求,尤其是在肿瘤研究和遗传病检测的应用方面。纳昂达的 MDI 方案进一步扩增至768 种 Index,以期满足更多用途需求。
表 2: MDI 设计原则
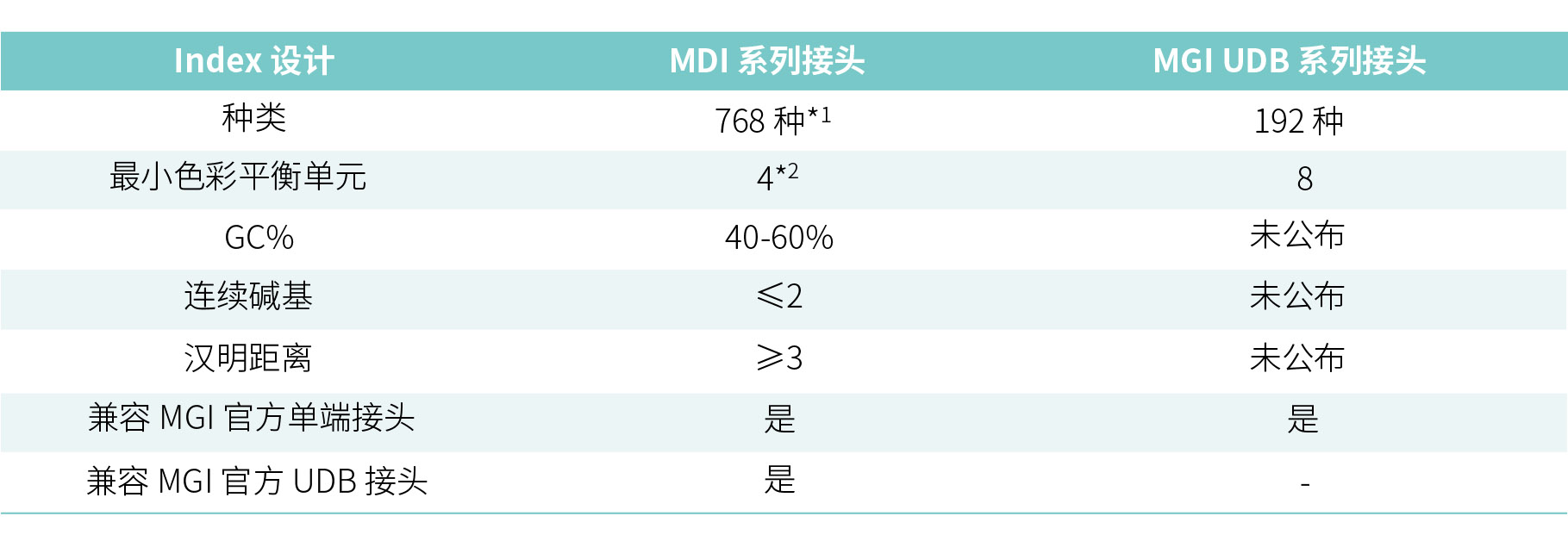
* 1-384已上市,385-768暂未上市销售;
* MDI 49-768的最小平衡单元为4,MDI 1-48的最小平衡单元为8。
设计时如使用最小色彩平衡为 8,则其最佳使用方式不得不要求每 lane 至少混合 8 种 Index,以确保平衡。而纳昂达的 MDI 序列设计的最小色彩平衡单元为 4,即 ≥4 个样本时,可连续使用。在大多数情形下,此种设计对使用方式更为友好。

图 1. A. NadPrep® DNA Library Preparation Module (for MGI) 分别搭配 NadPrep® Universal Adapter (MDI) Module (for MGI) 和 NadPrep® BMI Adapter (MDI) Module (for MGI) 构建文库的工作流程。
均一的文库和数据产出
MDI 系列方案,均通过独立扩增效率验证,以保证文库的均一产出(图 2A)。同时,多文库混合测序时,各 Index 间的数据有效拆分率也基本一致,以保证数据产出的均一性(图 2B)。

图 2. MDI 表现示例。 A. 96 个 MDI 类型文库产量。100 ng 片段化 gDNA,按 NadPrep® DNA Library Preparation Module (for MGI) 说明书操作,分别使用 MDI-97~MDI-192,扩增 5 个循环,平均文库产出均 >1000 ng;B. 96 个 MDI 类型文库的数据有效拆分率。测序模式:MGISEQ-2000,PE100。
有效提升数据准确度
为评估 MDI 的整体准确度和标签错配情况,我们构建多个 WGS 样本文库模拟混合并分 lane 测序。12、32 和 96 个 MDI 文库的数据拆分结果显示:所有 MDI 均可正确拆分,且比例 >99.9%(图 3)。使用双端匹配的 Index 拆分可大大减少样本读取的标签错配(Index 1 和 Index 2 匹配至不同的 MDI )。MDI 的标签错配率极低,可有效提升数据准确度,搭配更高的测序通量,可让临床研究更简单、有效的同时进一步降低成本。
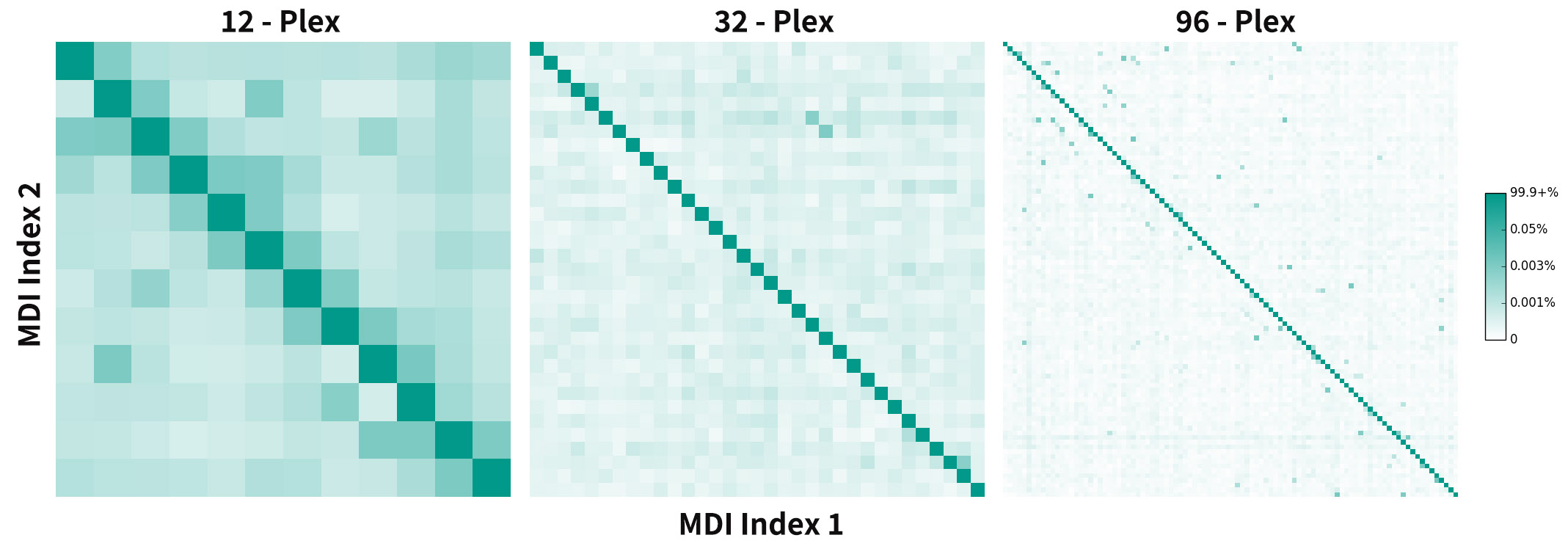
图3. MDI文库的数据串扰分析。 96个不同的MDI WGS文库,分别将12、32、96个文库混合,分lane测序。测序模式:MGISEQ-2000,PE100。
低频突变分析
MDI 系列方案通过与截短型接头组合可实现通用文库和分子标签文库构建,适用于不同的应用场景。分子标签文库经靶向捕获分析可用于血浆游离核酸的突变位点的检测、尿液游离核酸突变位点的检测、石蜡样本超低频突变检测等,具体可参阅纳昂达科技微信公众号文章“分子标签靶向捕获应用示例”。
表 3. MDI 搭配不同类型接头应用场景

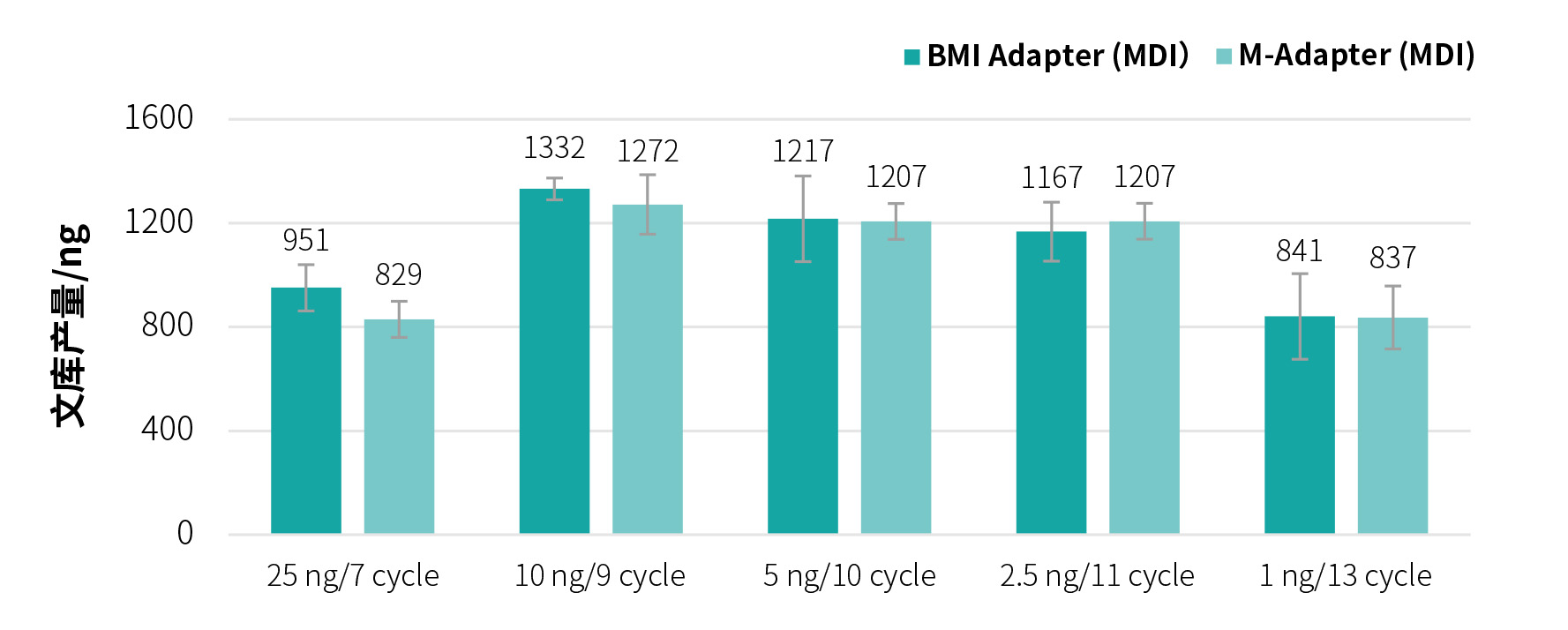
图 4.MDI 搭配 BMI Adapter 和 M-Adapter 的产量一致。
注: NadPrep® DNA Library Preparation Module (for MGI) 分别搭配 NadPrep® Universal Adapter (MDI) Module (for MGI) 和 NadPrep® BMI Adapter (MDI) Module (for MGI) 构建文库,样本为血浆提取的 cfDNA。
兼容并蓄
为方便用户使用,MDI 支持与 MGI UDB 接头混合上机。同样,MDI 也可兼容 MGI 单端接头使用。具体混合方案,详询 support@njnad.com。
参考文献
[1]https://www.illumina.com.cn/content/dam/illuminamarketing/documents/products/whitepapers/index-hopping-white-paper-770-2017-004.pdf?linkId=36607862.
[2] Li Q, Zhao X, Zhang W, et al. Reliable multiplex sequencing with rare index mis-assignment on DNB-based NGS platform[J]. BMC genomics, 2019, 20(1): 1-13.
[3] MacConaill L E, Burns R T, Nag A, et al. Unique, dual-indexed sequencing adapters with UMIs effectively eliminate index cross-talk and significantly improve sensitivity of massively parallel sequencing[J]. BMC genomics, 2018, 19(1): 1-10.







