ThalaScan:一站式地贫检测生信分析流程全新上线!
01 背景
地中海贫血 (以下简称为地贫) 是一种常见的单基因疾病,由 α 或 β 珠蛋白基因的致病性变异引发,这些变异导致 α 和 β 珠蛋白链之间的平衡失调。传统的地贫 (以及其他血红蛋白病) 的检测方法首先进行血液学测试,例如测定平均红细胞血红蛋白 (Hb) 和平均红细胞体积等指标,接着进行 Hb 电泳,最后通过基因测试来明确具体的致病变异。尽管在大多数情况下这一流程效果显著,但却需要耗费大量实验室工作和根据具体情况逐步制定决策,这使得大规模检测变得颇为繁琐。在某些罕见情况下,一些疾病突变可能不会呈现出血液学上的阳性特征,导致可能存在假阴性的血液学筛查情况。
珠蛋白基因的新发突变或复杂结构变化是遗传咨询和产前诊断领域的主要挑战。目前,为了预防地贫等相关疾病,携带者筛查和产前检测被视为首选的防护措施。传统的突变检测方法需要使用多种技术来分析各种类型的基因突变。然而,基于靶向富集的 NGS 技术能够显著提高突变检测的效率。相较于多重扩增子,探针杂交捕获更为高效,其具有以下优势:① 可检测到常见和不常见的突变;② 在大规模人群筛查中更具成本效益;③ 能够降低患者突变被误诊或漏诊的风险。
α-珠蛋白基因簇位于染色体 16p13.3 区域,该区域具备一系列独特特征,包括同源序列、散在重复序列以及各种类型的遗传变异。由于这种序列的同源性,NGS 测序的片段通常会映射到多个不同位置,而不是唯一的位置。这不仅会造成点突变和插入/缺失突变 (InDels) 的漏检,还会导致测序深度的不准确性,然而测序深度对于检测结构变异 (SVs) 是至关重要的。此外,在地贫流行地区,复合型结构变异的出现相当常见。例如,-- SEA/–α 3.7 是一种导致 Hb H 病的复杂杂合基因型,在中国香港和其他一些东南亚地区广泛存在。因此,利用生信分析工具准确检测这些复杂基因型对于全面实施地贫分子诊断和携带者筛查至关重要。
地贫生信分析流程 ThalaScan
在今年 1 月,纳昂达推出了全新的血红蛋白基因检测综合性 Panel——HGBP Panel v1.0。该 Panel 靶向人类血红蛋白编码基因、临近的基因组区域以及调节基因,共覆盖 160 Kb 基因组区域,可同时富集人类血红蛋白相关的多种变异信息,详情点击“新品 HGBP Panel | 异常血红蛋白、地中海贫血等遗传性血红蛋白疾病定制方案”。为了更好地与 HGBP Panel 的检测相配合,纳昂达精心研发了一项专门针对地贫检测的一站式生物信息学分析流程,即 ThalaScan,主要功能包括检测 Hb 区域中的点突变、InDels 以及 SVs,同时还能够检测位于调节区域的地贫致病变异和已知的疾病修饰因子。ThalaScan 的验证基于标准品数据和先前从临床实验室确认的具有珠蛋白基因变异的靶向测序数据,确保了其高度灵敏和特异性。
在本文中,我们将重点展示 ThalaScan 应用于拷贝数变异检测的结果。
02 材料与方法
2.1 样本来源
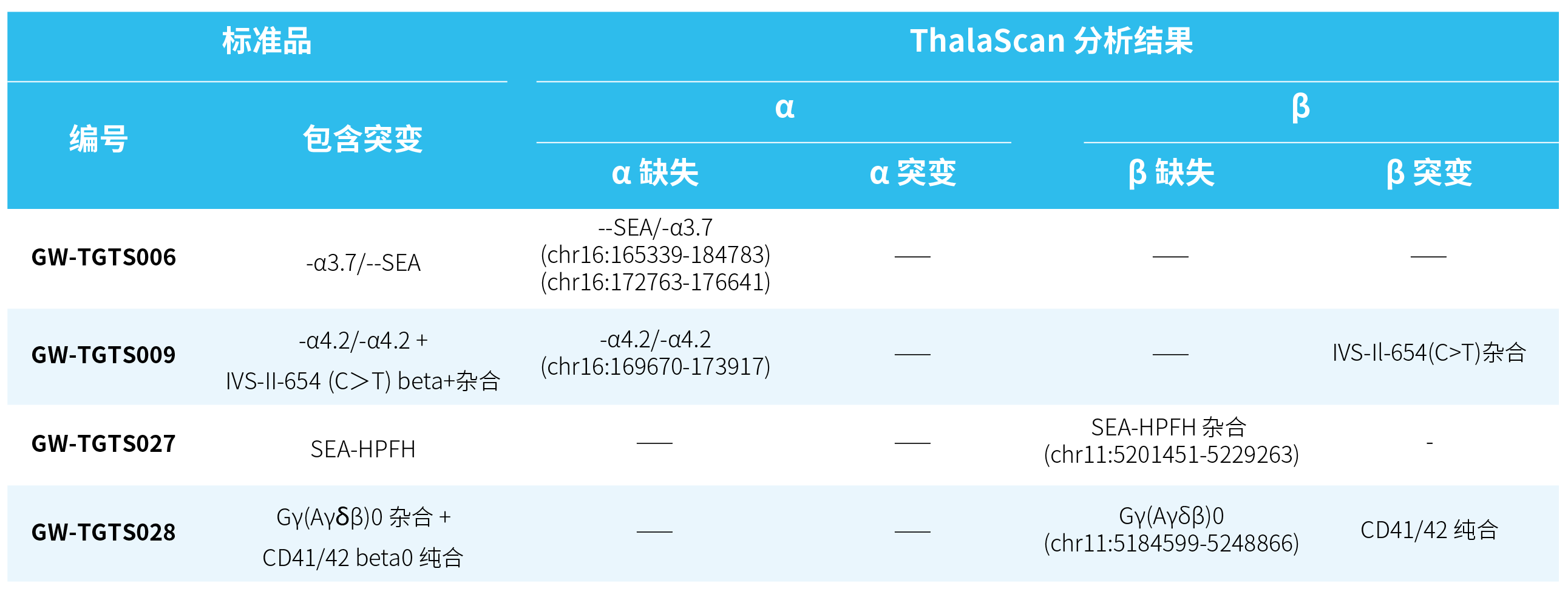
样本来源于 4 例地贫 gDNA 标准品 (Genewell) 和若干临床真实样本。4 例标准品的基因突变如表 1. 所示:
表 1. 4 例地贫 gDNA 标准品的基因突变情况

2.2 捕获测序方案
50 ng gDNA 利用 NadPrep® DNA 通用型文库构建试剂盒 (for Illumina®) 建库,以 HGBP Panel v1.0 完成杂交捕获 (6 plex),Illumina Novaseq 6000 PE150 测序。
2.3 ThalaScan 拷贝数变异检测算法和流程
ThalaScan 采用改进的逐步回归算法 (Stepwise Regression Algorithm) 分析地贫样本的拷贝数变异。这一分析流程主要分成两个关键步骤:1)采用滑窗的方式对目标区域内的每个窗口 (bin) 进行矫正,并计算拷贝数;2)根据 softclip read 过滤后的断点位置,进行联合分析,以判断拷贝数变异发生的范围和绝对值。
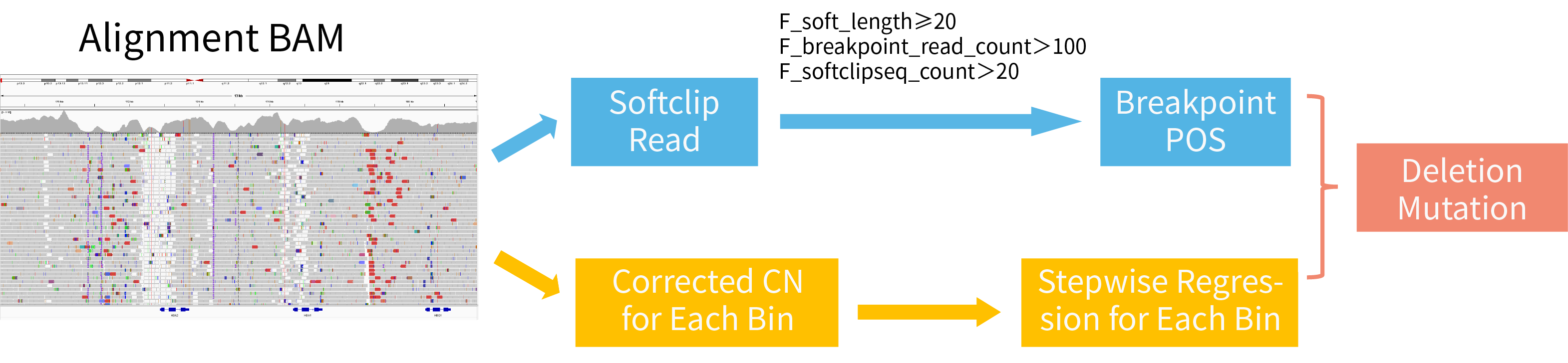
图 1. ThalaScan 拷贝数变异检测流程
03 拷贝数变异检测分析结果
3.1 标准品样本
样本 1:GW-TGTS006
首先将该样本的原始数据进行质控和比对分析,中靶率在 80% 以上,0.5x mean 在 90% 以上。经过比对后,生成的 BAM 文件在 IGV 中进行了可视化展示,如图 2. 所示:
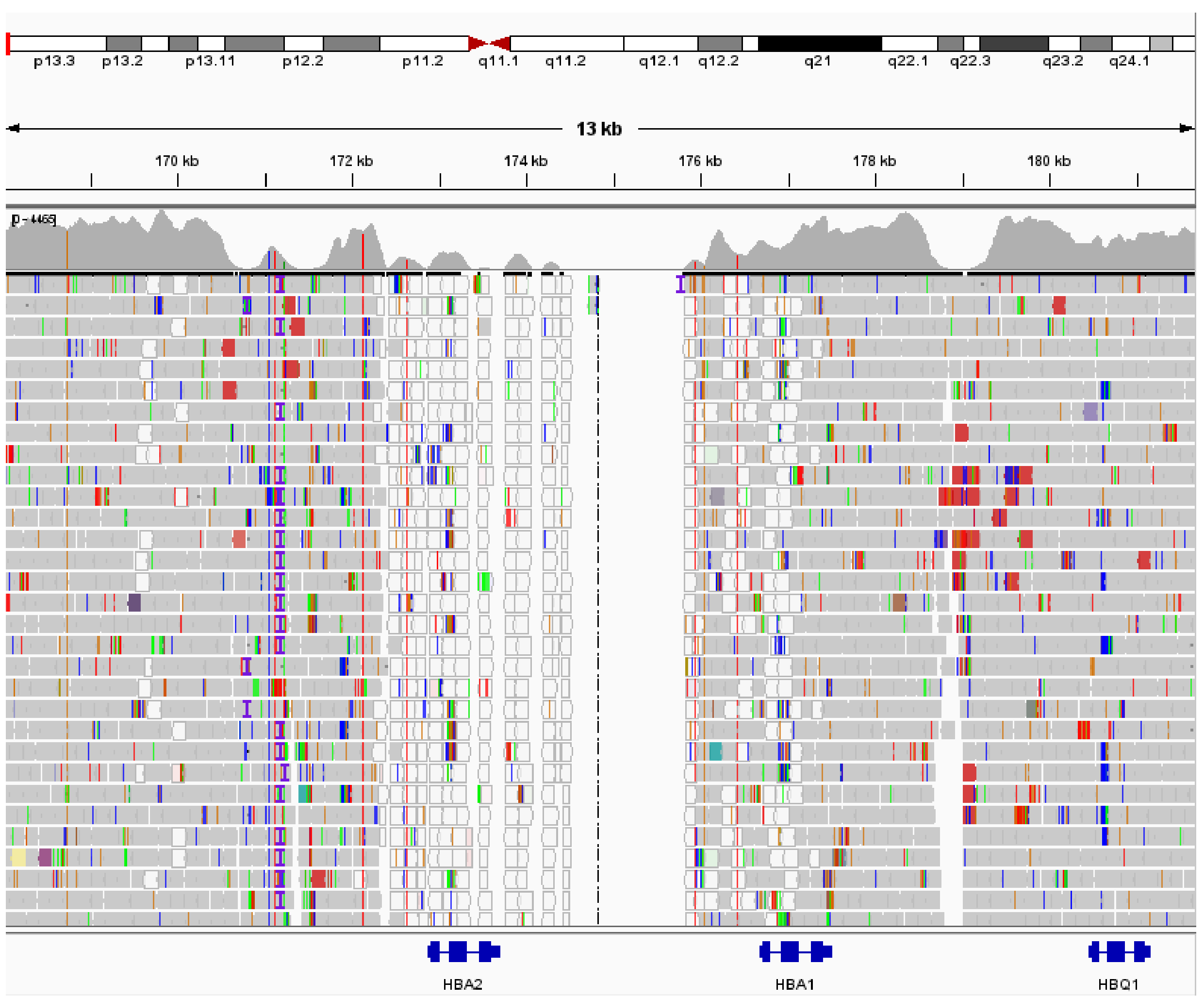
图 2. GW-TGTS006 HBA1 和 HBA2 基因 IGV 分布
通过 IGV 分布图,可以观察到 HBA1 和 HBA2 之间的间隔区发生了纯合缺失,而且 HBA2 覆盖的 read 的比对质量值很低 (这是由于 HBA1 和 HBA2 之间的同源性导致的)。然而,仅仅通过 IGV 展示很难准确判断拷贝数变异的范围和绝对 CN 值。为了更清晰地可视化拷贝数变异,本研究将目标区域的每个滑窗 bin 都看作是基因组上的单独位置坐标,并将经过矫正的 CN 值作为纵坐标,以图形的形式展示,具体如图 3. 所示。
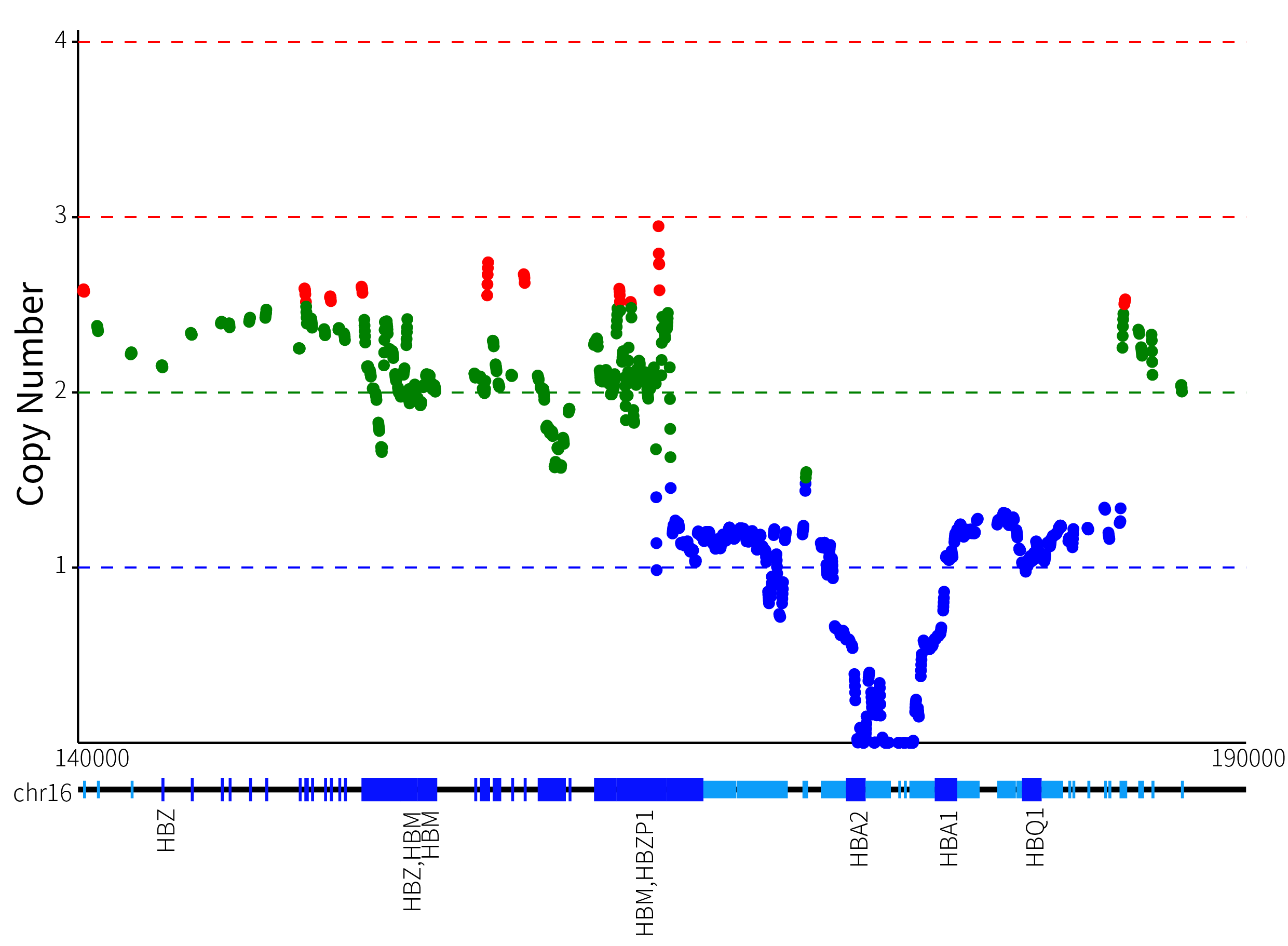
图 3. GW-TGTS006 Alpha 基因簇拷贝数可视化展示
从图 3. 的可视化展示中,可以清晰地看出,GW-TGTS006 的 α 基因簇发生了复合缺失突变,导致绝对 CN 值为 0 和 1 的缺失型变异。为了更加准确地确定该复合缺失发生的具体位置,我们采用了基于 softclip read 断点和逐步回归算法的方法,以图 4. 所示的方式来确定该样本中真正发生缺失变异的范围。
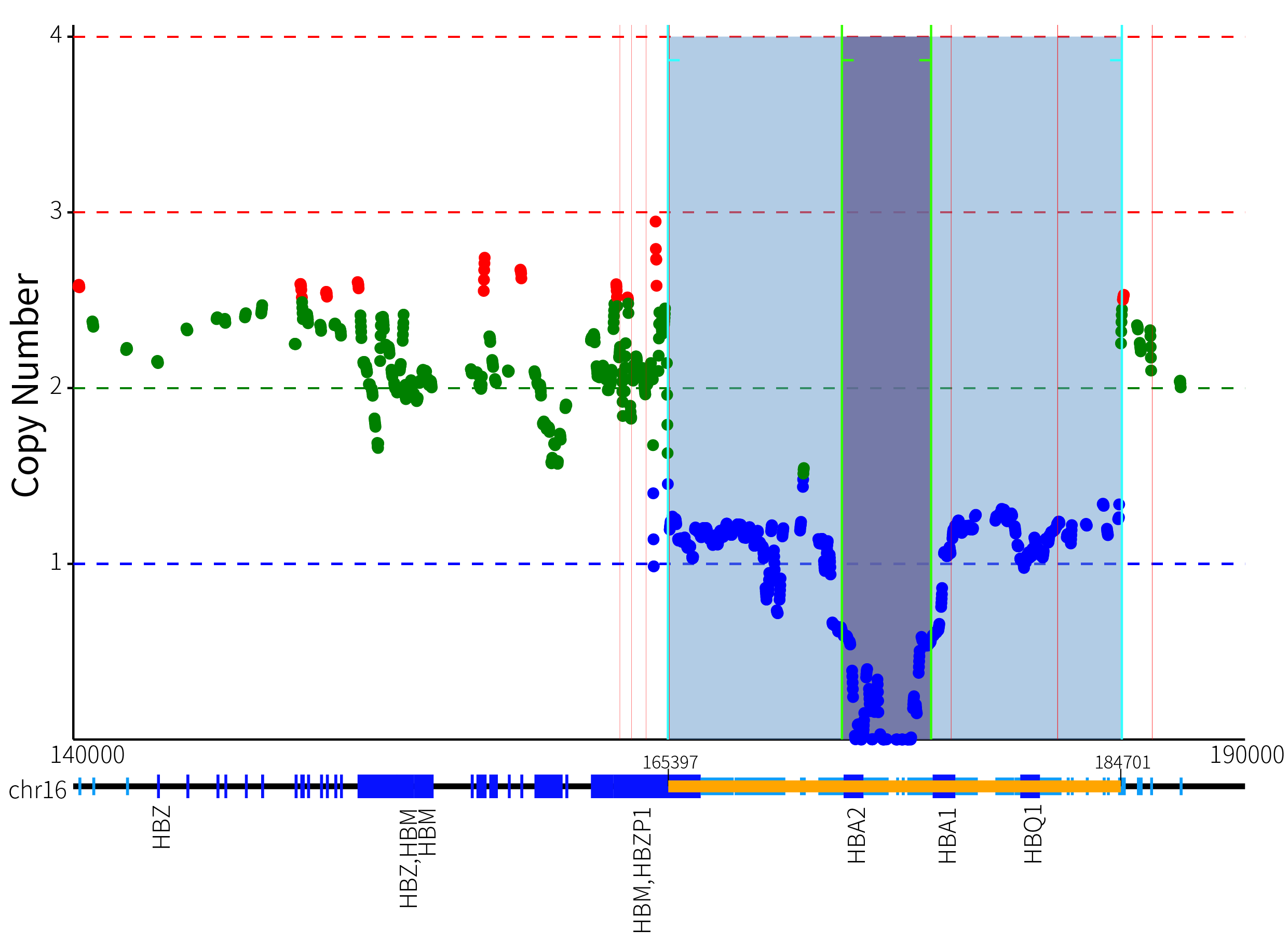
图 4. GW-TGTS006 复合突变断点位置
在图 4. 中,红色竖线标示了可能的缺失变异断点位置。基于 ThalaScan,通过逐步回归算法,我们确定了拷贝数变异 CN 值为 1 和 0 的四个断点位置,并用绿色竖线标记了这些位置。同时,我们用黄色条形框在基因组横坐标上标记了拷贝数变异的范围,清晰地显示该拷贝数变异覆盖了 HBQ1、HBA1、HBA2 以及部分 HBZP1-HBM 基因。通过将精确的断点位置与 HbVar 数据库进行比较,我们得出结论,该样本出现了 --sea/-a3.7 的复合缺失突变。
样本 2:GW-TGTS009
在本次研究中,我们同样将 GW-TGTS009 样本进行了质控和比对,并查看了其比对后的 BAM 文件在 IGV 中的分布和 Alpha 基因簇的可视化展示,如图 5. A 和图 5. B 所示。通过 IGV 分布图,可以观察到 HBA2 上游发生了纯合缺失,而且 HBA2 的覆盖以及部分 HBA1 的 read 的比对质量值很低 (这是由于 HBA1 和HBA2 之间的同源性造成的)。从图 5. B 的 Alpha 基因簇的可视化结果来看,HBA1 与 HBA2 之间的凹凸是由于 HBA1 和 HBA2 的同源错误比对引起的,这会导致对 CN 值的误判。实际上,HBA1 并没有发生任何缺失,而 HBA2 是纯合缺失。基于 ThalaScan,如图 5. C 所示,我们得出结论,该样本出现了 -a4.2/-a4.2 的纯合缺失突变。

图 5. A. GW-TGTS009 HBA1 和 HBA2 基因 IGV 分布;B. GW-TGTS009 Alpha 基因簇拷贝数可视化展示;C. GW-TGTS009 缺失突变断点位置。
样本 3:GW-TGTS027
在本次研究中,我们同样将 GW-TGTS027 样本进行了质控和比对,并查看了其比对后的 BAM 文件在 IGV 中的分布和 Beta 基因簇的可视化展示,如图 6. A 和图 6. B 所示。通过 IGV 分布图,可以观察到 HBB 上游发生了杂合性缺失,但由于 IGV 显示问题,无法直观展示所有的杂合性缺失位置。图 6. A 中红色箭头所示的部分表示未放置探针的区域,这也在一定程度上影响了后续拷贝数变异的范围展示。从图 6. B 的 Beta 基因簇的可视化结果来看,可以清晰地看出,杂合性缺失范围覆盖了 HBB 基因以及其上游区域。基于 ThalaScan,如图 6. C 所示,我们得出结论,该样本出现了 SEA-HPFH 的杂合缺失突变。

图 6. A. GW-TGTS027 HBB 基因 IGV 分布;B. GW-TGTS027 Beta 基因簇拷贝数可视化展示;C. GW-TGTS027 缺失突变断点位置。
样本 4:GW-TGTS028
在本次研究中,我们同样将 GW-TGTS028 样本进行了质控和比对,并查看了其 Beta 基因簇的可视化展示,如图 7. A 所示。由于该样本的 Beta 基因簇的杂合性缺失范围过大,IGV 无法进行有效的展示。而从图 7. A. 的 Beta 基因簇的可视化结果来看,可以清晰地看出,杂合性缺失范围覆盖了 HBB 基因及其上下游区域,包括了 HBD,HBBP1,HBG1 和 HBG2 等区域。基于 ThalaScan,如图 7. B 所示,我们得出结论,该样本出现了 Gγ(Aγδβ)0 的杂合缺失突变。

图 7. A. GW-TGTS027 Beta 基因簇拷贝数可视化展示;B. GW-TGTS028 缺失突变断点位置。
表 2. 标准品变异检测结果汇总
3.2 临床样本
真实临床样本进行了质控和比对,基于 ThalaScan,我们确定了 4 例临床样本真实发生缺失变异的范围,如图 8. 所示。
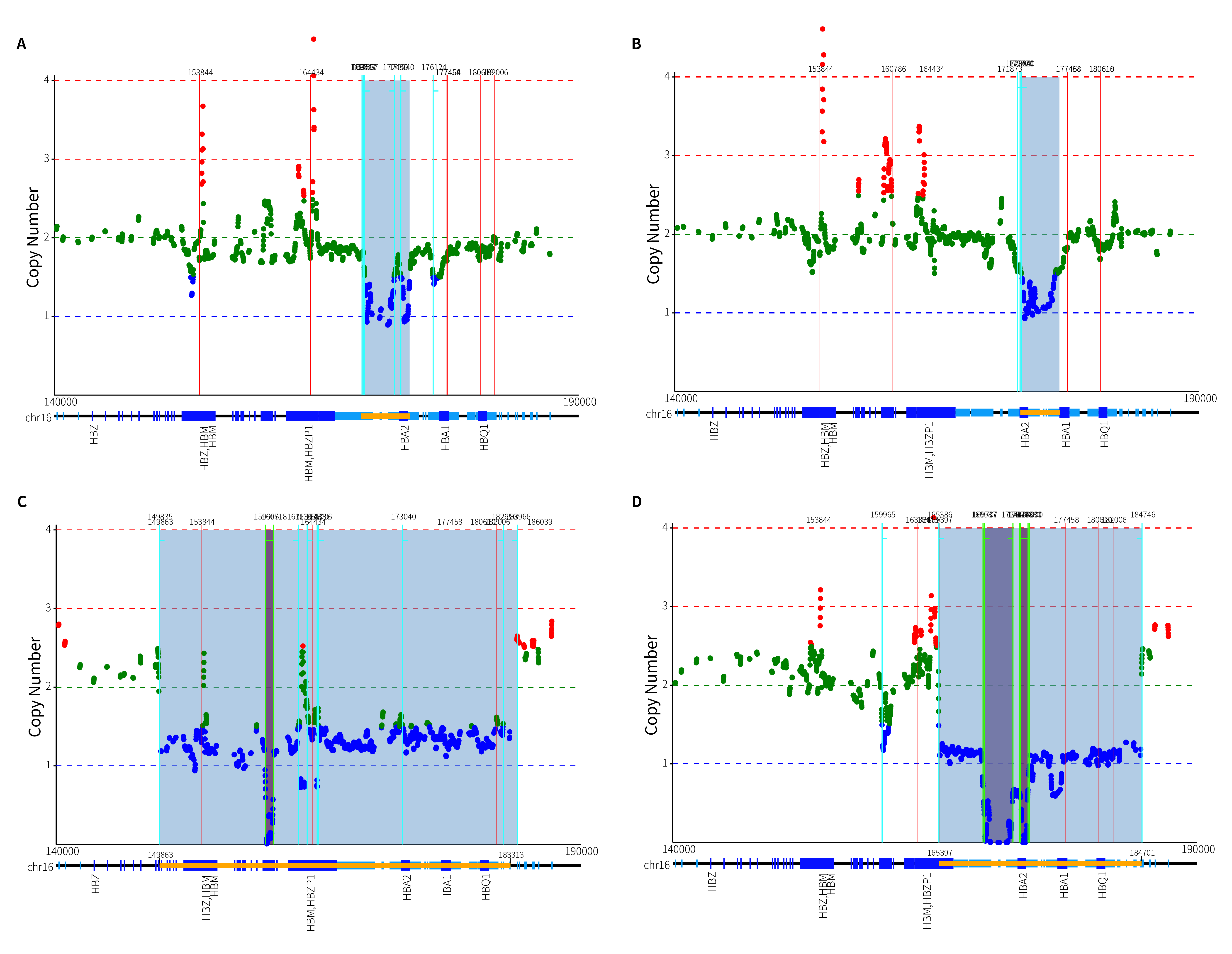
图 8. 4 例临床样本变异检测分析结果
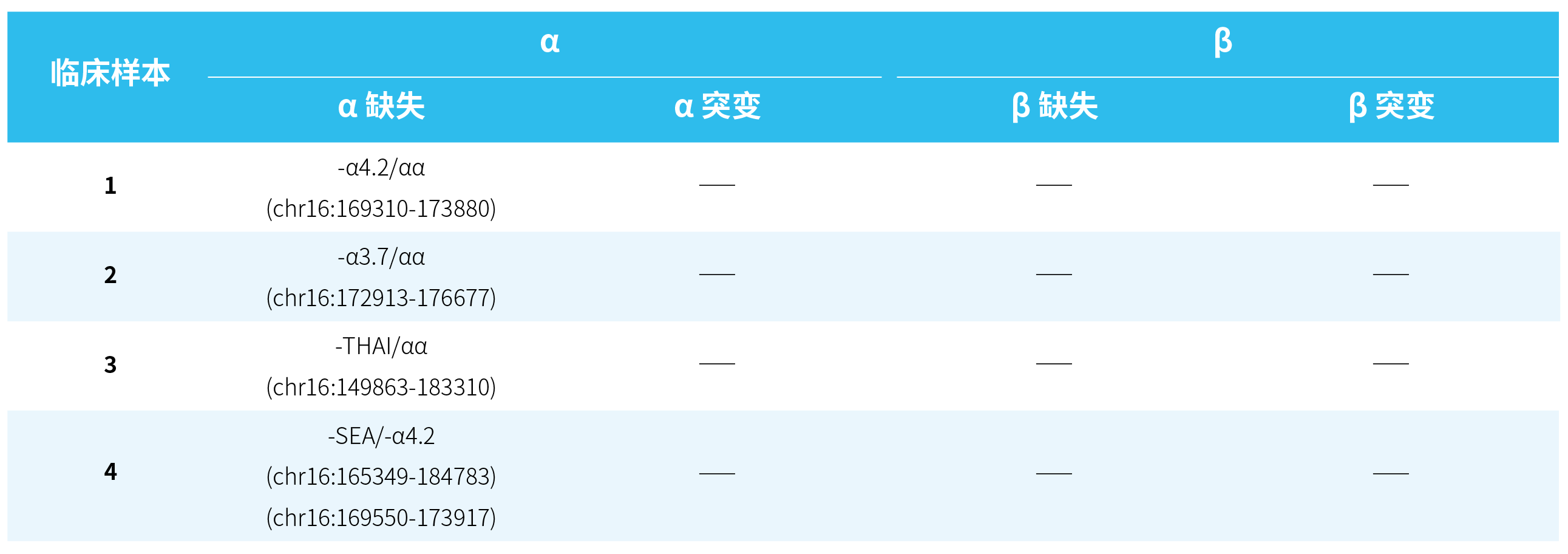
表 3. 4 例临床样本变异检测结果汇总
04 讨论
针对 Hb 区域的 NGS 测序具备检测几乎所有基因变异的潜力。然而,Hb 区域的序列复杂性给 NGS 数据分析带来了巨大挑战。由于生物信息学专业知识在临床实验室中尚未广泛传播,使得数据分析较难开展。
我们开发的 ThalaScan 算法流程,搭配相应的捕获方案,不仅能够检测已知的地贫 (通过自动化在 HbVar 数据库中查找检测到的已知变异),还能够检测新的 Hb 变异、疾病修饰因子以及导致其他血红蛋白病的变异。
本文中展示的地贫拷贝数变异分析数据,多基于已知变异类型。事实上,应用于真实临床样本时,比已知 Hb 基因区域更长的片段缺失并不罕见。ThalaScan 则通过对 HGBP Panel 中临近区域 SNP 位点同步分析来揭示这些新变异。如果在先证者的 SNP 位点违反了孟德尔遗传法则,还能进一步提升分析的准确度。
我们将陆续分享更多 ThalaScan 应用于真实临床样本中的检测结果,欢迎垂询,敬请期待。
参考文献
[1] Piel F B, Weatherall D J. The
α-thalassemias[J]. New England Journal of Medicine, 2014, 371(20): 1908-1916.
[2] Rund D, Rachmilewitz E.
Beta-thalassemia[J]. New England Journal of Medicine, 2014, 353(11):1135-46.
[3] Giardine B, Borg J, Viennas E, et al.
Updates of the HbVar database of human hemoglobin variants and thalassemia
mutations[J]. Nucleic acids research, 2014, 42(D1): D1063-D1069.
[4] Giardine B, Borg J, Higgs D R, et al.
Systematic documentation and analysis of human genetic variation in
hemoglobinopathies using the microattribution approach[J]. Nature genetics,
2011, 43(4): 295-301.