乳腺癌 NAC 预后效果及复发 Biomarker 新探索
01 背景
乳腺癌是中国女性最常见的癌症,每年新增病例达 30.4 万例,死亡人数达 7 万人[1]。传统的乳腺癌治疗方式预后效果有限,而新辅助化疗 (NAC) 自 2015 年以来已成为局部晚期乳腺癌的标准治疗方法[2],其预后效果较传统方法具有显著提升[3]。
循环肿瘤 DNA (ctDNA) 测序作为一种非侵入性的检测方法, 近年来在乳腺癌的早期筛查、诊断、治疗和预后方面得到了广泛应用[4-6]。ctDNA 检测和分析的一个潜在优势在于其能够近乎实时地监测患者对治疗的反应,有助于迅速了解特定治疗方案的有效性,从而使得治疗方案能够及时调整。然而,目前对于 ctDNA 在评估 NAC 乳腺癌治疗中的价值仍然存在一定得不明确性。
重庆陆军医科大学大坪医院乳腺甲状腺外科发表的文章 “Analysis of
Circulating Tumor DNA to Predict Neoadjuvant Therapy Effectiveness and Breast
Cancer Recurrence”,首次利用靶向捕获测序技术评估了 NAC 前后患者 ctDNA 中的突变特征,以及手术期间组织中的突变。该研究突显了在不同时间点进行 ctDNA 分析以评估肿瘤进展和治疗效果,以及预测乳腺癌复发的潜力。
研究人员通过靶向捕获测序对患者 ctDNA 中 727 个乳腺癌相关的基因进行了分析,其中所采用的定制化 Panel 源自纳昂达。
02 研究方法
样本来源:研究人员招募了 31 名乳腺癌患者,并采集了所有 31 名患者 NAC 前 (C1) 的外周血样本,手术期间 (T) 的肿瘤组织样本,以及 25 名患者手术后 6 个月 (C2) 的外周血样本。
靶向捕获测序:利用患者外周血样本提取的 cfDNA 和 gDNA 以及组织样本中提取的 tDNA 分别进行文库制备,将制备好的 DNA 文库与靶向 727 个基因的定制化 Panel (Nanodigmbio) 进行杂交捕获。
生信分析:

03 结果
3.1 靶向捕获测序和遗传信息概述
研究人员对所有样本 (来自 31 名患者的 87 个样本) 应用了纳昂达定制化 Panel,以实现靶向捕获测序。其中,C1、T 和 C2 样本的平均捕获效率分别为 72.89%、74.98% 和 72.80%,而有效目标深度分别为 1010.19x、1071.61x 和 925.15x。
总体而言,研究人员在 C1、T 和 C2 样本中分别鉴定到 159、271 和 70 个体细胞突变。在 31 个 C1 样本中均检测到的体细胞突变为 30 个,而在 25 个 C2 样本中为 12 个。与 T 样本相比,T 样本中每个样本检测到的平均突变数为 8.7,而 C1 样本中每个样本的突变数明显较低 (5.1)。此外,C1、T 和 C2 样本中分别检测到了 10、26 和 10 个体细胞热点突变,而这些体细胞热点突变都属于一种类型,即 KMT2C
c.5053G>T。这些结果揭示了我们研究早期和中期的血液和组织样本之间存在较高的异质性[7]。考虑到所有 87 个样本,最常见的突变基因是 KMT2C (20.69%)、PIK3CA (18.39%) 和TP53 (18.39%) (图 1.),而正是这些差异明显导致了不同时间点样本的异质性。
除了体细胞突变外,研究人员还在 31 名乳腺癌患者中鉴定到 6 个种系突变 (19.35%),涉及 BRCA2、CHEK2 和 MUTYH。通过单变量和多变量分析,这些突变被确定与病理完全缓解 (pCR) 和复发无统计学相关性。然而在鉴定出的 6 个种系突变中,有 5 个出现在乳腺癌的激素受体阳性亚型中。
图 1. 结果表明,研究人员总共发现了 131 个 CNV。在组织样本中,有 35 个基因具有 CNV,在复发患者中,C2 样本中没有发现 CNV。基于此,CNV 可能不是早期乳腺癌复发监测的良好生物标志物。

图 1. 由 31 位患者的 87 个样本组成的突变图谱,包括 Top 20 突变基因 (SNV + InDels)。
3.2 ctDNA KMT2C c.5053G>T 与乳腺癌复发的关联
已有研究证明,淋巴结状态和 pCR 可作为乳腺癌复发的预测因子。为了探讨术后血液中的 ctDNA 是否也可作为新辅助治疗后复发的预测因子,研究人员对 C1、T 和 C2 样本中的体细胞突变进行了比较,在 8 个 C1 样本、3 个 T 样本和 7 个 C2 样本中检测到了 KMT2C 突变。生存分析 (Kaplan-Meier) 显示 ctDNA KTM2C c. 5053G>T 与术后乳腺癌复发显著相关 (p =
0.0014) (图 2.)。尽管在 P5 和 P18 中 KMT2C c.5053G>T 的等位频率下降,但仍然相对较高 (图 3. A-D)。这些发现表明 ctDNA 突变可以作为新辅助治疗后乳腺癌复发的潜在预测因子。而与 CA 15-3、CA 12-5 和 CEA 的结果相冲突表明,传统的肿瘤生物标志物无法满足早期肿瘤复发检测的需求。
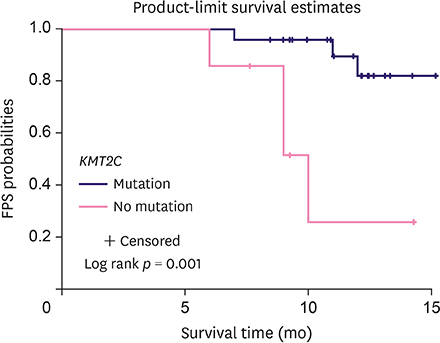
图 2. KMT2C 突变与复发时间 (月) 的 Kaplan-Meier 曲线。
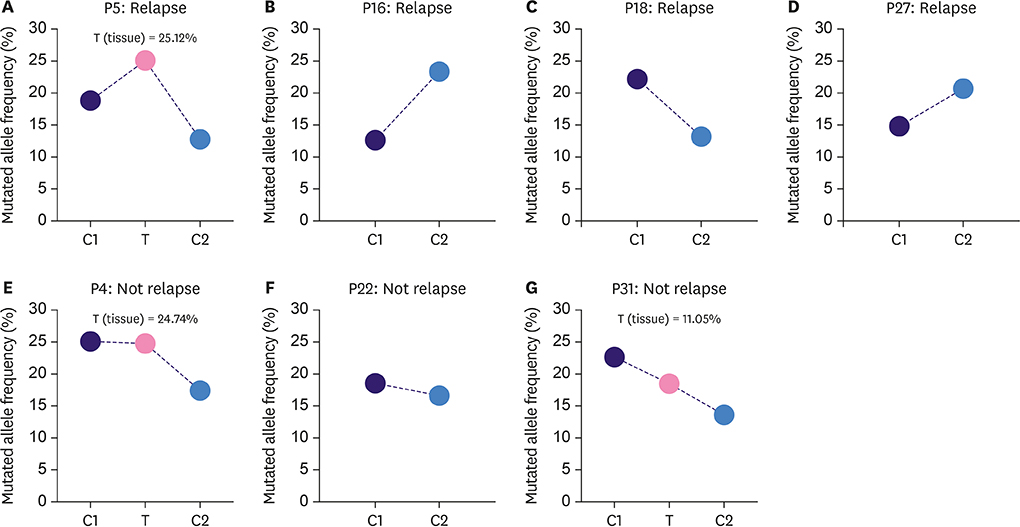
图 3. 不同时间点 7 名患者中 KMT2C c.5053G>T 突变等位基因频率。
注:对于组织样本,在患者 P5、P4 和 P31 样本中检测到 KMT2C 突变,而在患者 P16、P18、P27 和 P22 样本中未检测到。
最后,研究人员对 C1 外周血样本中的肿瘤突变负荷 (TMB) 和突变情况进行了分析。在 C1 样本中鉴定的 8 个 KMT2C 突变中,4 名患者在新辅助治疗后表现出病情稳定 (SD),而其余 4 名患者则达到了 pCR。基于 χ2 检验,KMT2C 与新辅助治疗效果之间的关联并不显著 (p > 0.05),且 TMB 的有效性也不显著。然而,研究人员发现一些具有或没有 KMT2C 突变的 SD 患者具有相对较高的 TMB,这表明在大型队列中,通过 KMT2C 与高 TMB 的联合可能可以预测新辅助治疗的有效性。
04 总结
据了解,这项研究是首个探讨中国乳腺癌患者新辅助治疗后 ctDNA 与预后之间关系的研究。通过靶向测序发现了 KMT2C 突变在乳腺癌患者中的高发生率。然而,研究结果显示,KMT2C 与新辅助治疗效果之间的关联并不显著,无论是 CNV 还是 TMB 都不是新辅助治疗和乳腺癌复发的可靠预测指标。该研究依然存在较多的局限性和进一步探究的空间,可能需要考虑从延长随访时间、增加样本量以及采用更新鲜的组织样本等方法深入探讨。
在癌症预后效果、诊断和复发的新 biomarker 探索过程中,靶向捕获测序一直是研究人员的得力助手。纳昂达一直坚持以高标准和高质量为核心,保持初心,致力于为精准医疗服务提供不懈支持!
05 订购信息

NAD Probes 定制探针
NAD Probes 定制探针方案是纳昂达倾力打造的一站式服务,涵盖从探针设计、生产合成、质检及优化的全流程。通过严格品质控制和优化的 NGS QC 服务,可为客户提供高质量定制化杂交捕获解决方案,覆盖应用类型除常规突变检测外,还包括:甲基化捕获、RNA 捕获、病毒捕获等。
每条探针序列明确,浓度已知
ISO13485 体系生产标准,结果均一稳定
覆盖度均一性好,GC 偏好性小
支持 spike-in 至其它 NadPanel 或定制 Panel
支持多样本混合捕获

XCapert 探针在线设计平台
XCapert 为纳昂达推出国内唯一端到端 (一站式) 靶向捕获解决方案
完全自动化线上探针设计和个性化线下沟通设计,可在线一键操作,便捷高效
基于 >100 万条探针优化算法,不同格式文件可置入同一压缩包同时上传,轻松批量设计探针
外部 API 接口全新加入,探针设计更灵活

参考文献
[1] Zheng RS, Sun KX, Zhang SW, Zeng HM,
Zou XN, Chen R, et al. Report of cancer epidemiology in China, 2015. Zhonghua
Zhong Liu Za Zhi 2019;41:19-28.
[2] Haddad TC, Goetz MP. Landscape of
neoadjuvant therapy for breast cancer. Ann Surg Oncol 2015;22:1408-15.
[3] Esserman LJ, Berry DA, DeMichele A,
Carey L, Davis SE, Buxton M, et al. Pathologic complete response predicts
recurrence-free survival more effectively by cancer subset: results from the
I-SPY 1 TRIAL--CALGB 150007/150012, ACRIN 6657. J Clin Oncol 2012;30:3242-9.
[4] Buono G, Gerratana L, Bulfoni M,
Provinciali N, Basile D, Giuliano M, et al. Circulating tumor DNA analysis in
breast cancer: is it ready for prime-time? Cancer Treat Rev 2019;73:73-83.
[5] Zhang X, Zhao W, Wei W, You Z, Ou X,
Sun M, et al. Parallel analyses of somatic mutations in plasma circulating
tumor DNA (ctDNA) and matched tumor tissues in early-stage breast cancer. Clin
Cancer Res 2019;25:6546-53.
[6] Coombes RC, Page K, Salari R, Hastings
RK, Armstrong A, Ahmed S, et al. Personalized detection of circulating tumor
DNA antedates breast cancer metastatic recurrence. Clin Cancer Res
2019;25:4255-63.
[7] Shatsky R, Parker BA, Bui NQ, Helsten
T, Schwab RB, Boles SG, et al. Next-generation sequencing of tissue and
circulating tumor DNA: the UC San Diego Moores Center for personalized cancer
therapy experience with breast malignancies. Mol Cancer Ther 2019;18:1001-11.