填补技术空白!IF82.9!新一代三合一无创产前综合性筛查临床研究成果发表
01 背景
传统的产前筛查依赖于超声成像、母体血清学标志物,存在检出率低,假阳性高等局限性,基于 NGS 的无创产前检测 (NIPT) 是当下最全面和应用广泛的方法,可检测染色体非整倍体和微缺失/微重复综合征。出生缺陷是在宫内、出生时或婴儿后期可能出现的结构或功能异常[1]。在活产儿中,出生缺陷的患病率约为 2~4%[2]。在所有出生缺陷中,单基因病占比为 7.5%~12%,单基因病防控理应是出生缺陷防控体系中的重要一环[3]。但是,受传统技术的限制,目前的 NIPT 不包含单基因病,把单基因病纳入目前的筛查体系亟待技术突破。
为克服这一局限性,黄荷凤院士团队于 2022 年 10 月 13 日于 Cell
Discovery 上发表了可同步筛查染色体非整倍体、染色体微缺失综合征和单基因显性遗传病的综合性无创产前筛查技术——协同等位基因靶向富集测序 (coordinative allele-aware
target enrichment sequencing, COATE-seq) 的方法学和回顾性临床研究论文[4],通过检测孕妇血浆中的 cfDNA,全面筛查胎儿的不同类型的遗传变异。研究表明分析检测灵敏度 100%,特异性 99.3%。然而在真实的临床场景下,该综合筛查技术的实际临床性能仍有待阐明。
2024 年 01 月 22 日,复旦大学生殖与发育研究院黄荷凤院士携复旦大学和浙江大学等单位的研究人员在国际顶刊 Nature Medicine (IF = 82.9) 发表了题为 “Prospective prenatal cell-free DNA screening for genetic conditions of
heterogenous etiologies” 的最新临床研究结果。这项前瞻性、多中心的观察性研究,采用创新性的综合性无创产前筛查技术 (cfDNA 综合筛查),共计纳入 1,090 名高风险孕妇。cfDNA 综合筛查技术检测敏感性达 98.5%,特异性为 99.3%。并且相比于传统的只包括染色体疾病的 NIPT 筛查,提高对致病性遗传变异的检出率达到 60.7%;验证了其在产前同时筛查胎儿染色体病、微缺失微重复综合征和单基因病的临床有效性。其中测序方案的 cfDNA 文库构建试剂全部来自纳昂达 (Nanodigmbio)。

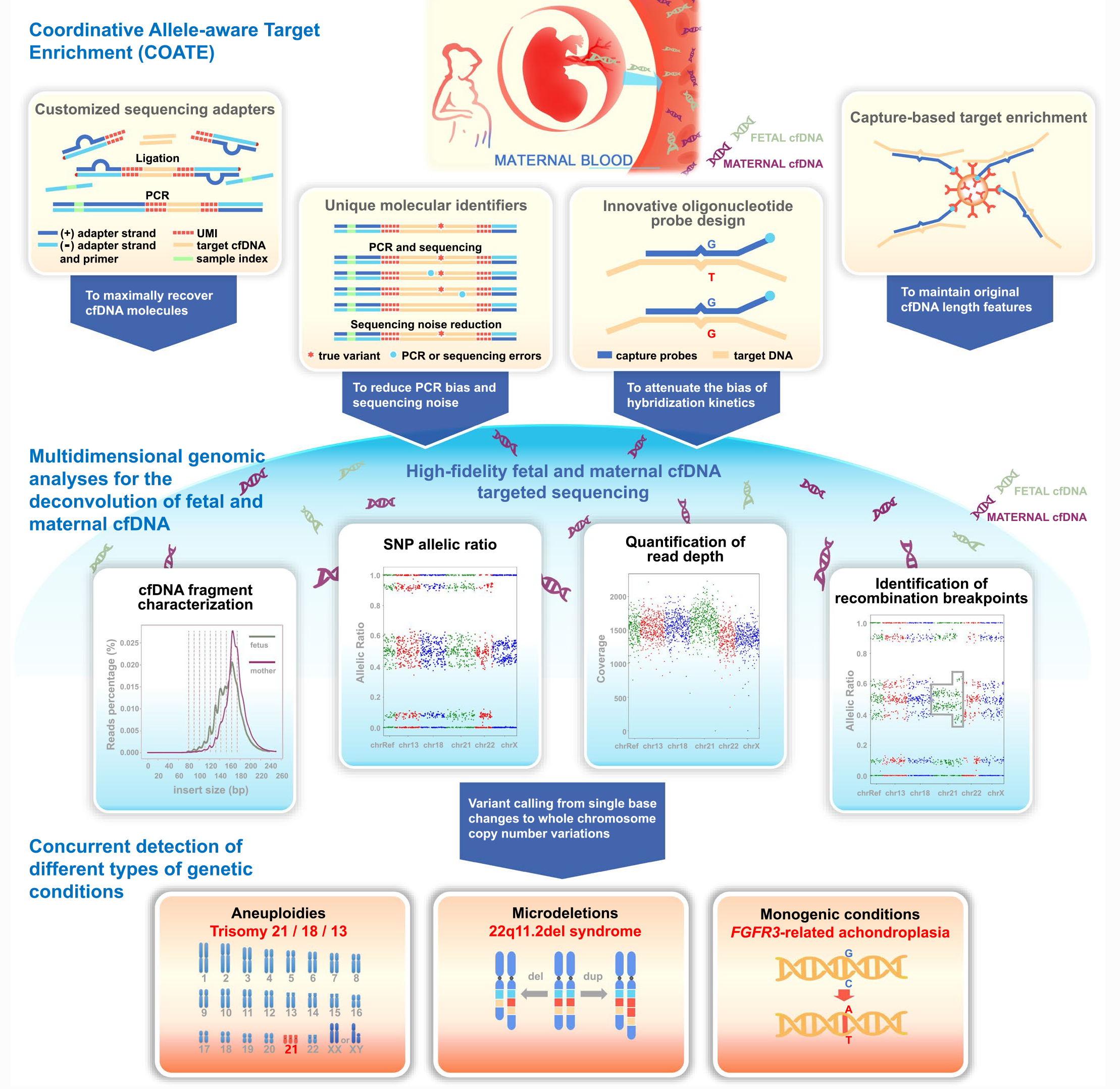
图 1. 新一代产前 cfDNA 综合筛查技术流程
02 研究方法
方案设计:该研究纳入了在 2021 年 04 月 24 日至 2022 年 09 月 10 日期间,来自中国不同省份的三家医院的 1,191 名孕妇 (1,090 名合格参与者),所有参与者都进行了无创产前三合一综合性筛查 (NIPT2 Pro) ,包括分析 7 种常见染色体非整倍体、9 种微缺失综合征以及与显性单基因病相关的 75 个基因。遗传诊断通过对绒毛取样、羊水穿刺、胎儿停育产物等样本的分析进行。
文库构建:该研究中的综合性产前 cfDNA 筛查采用一种称为 COATE-seq 的靶向测序方法[4]。母体血浆 cfDNA 使用 NadPrep cfDNA Library Preparation
Kit (for MGI) (Nanodigmbio) 搭配分子标签接头 (BMI) 进行文库构建,按照说明书完成靶向富集,测序模式为 MGISEQ-2000,PE100。
单基因变异、微缺失和非整倍体的 cfDNA 分析:单基因序列变异检测的最低测序深度阈值为 200x。所有样本中感兴趣基因的平均覆盖度为 1,387x,平均超过 99.3% 的靶区域符合最低覆盖度要求 (200x)。变异调用的主要算法是 Mutect2,使用两种额外的过滤方法即等位基因计数分布 (ACD) 和胎儿-母体插入片段大小分布 (FMID),来识别胎儿单基因变异。
03 结果
3.1 产前 cfDNA 三合一综合性筛查的临床有效性
该研究共入组 1,191 名孕妇 (图 1. )。其中,101 例被排除在外,包括 71 例没有诊断结果、15 例母体存在目标基因组区域变异、8 例未通过测序深度质控要求以及 7 例因多胎妊娠或样本污染未通过单胎质控要求。最终队列 (N = 1,090) 中所有孕妇的胎儿均被评估为遗传病高风险,包括 876 例 (80.4%) 胎儿超声异常,116 例 (10.6%) 母体血清学筛查结果异常,86 例 (7.9%) 标准 cfDNA (NIPT 或NIPT Plus) 染色体筛查结果为高风险,12 例 (1.1%) 曾有不良妊娠史,提示胎儿患遗传病风险增加。在最终队列的 1,090 例中,综合性产前cfDNA 筛查在 135 名 (12.4%) 孕妇中检出了致病性遗传变异,包括 89 例非整倍体、9 例微缺失和 37 例单基因疾病,并通过诊断检测加以确认。
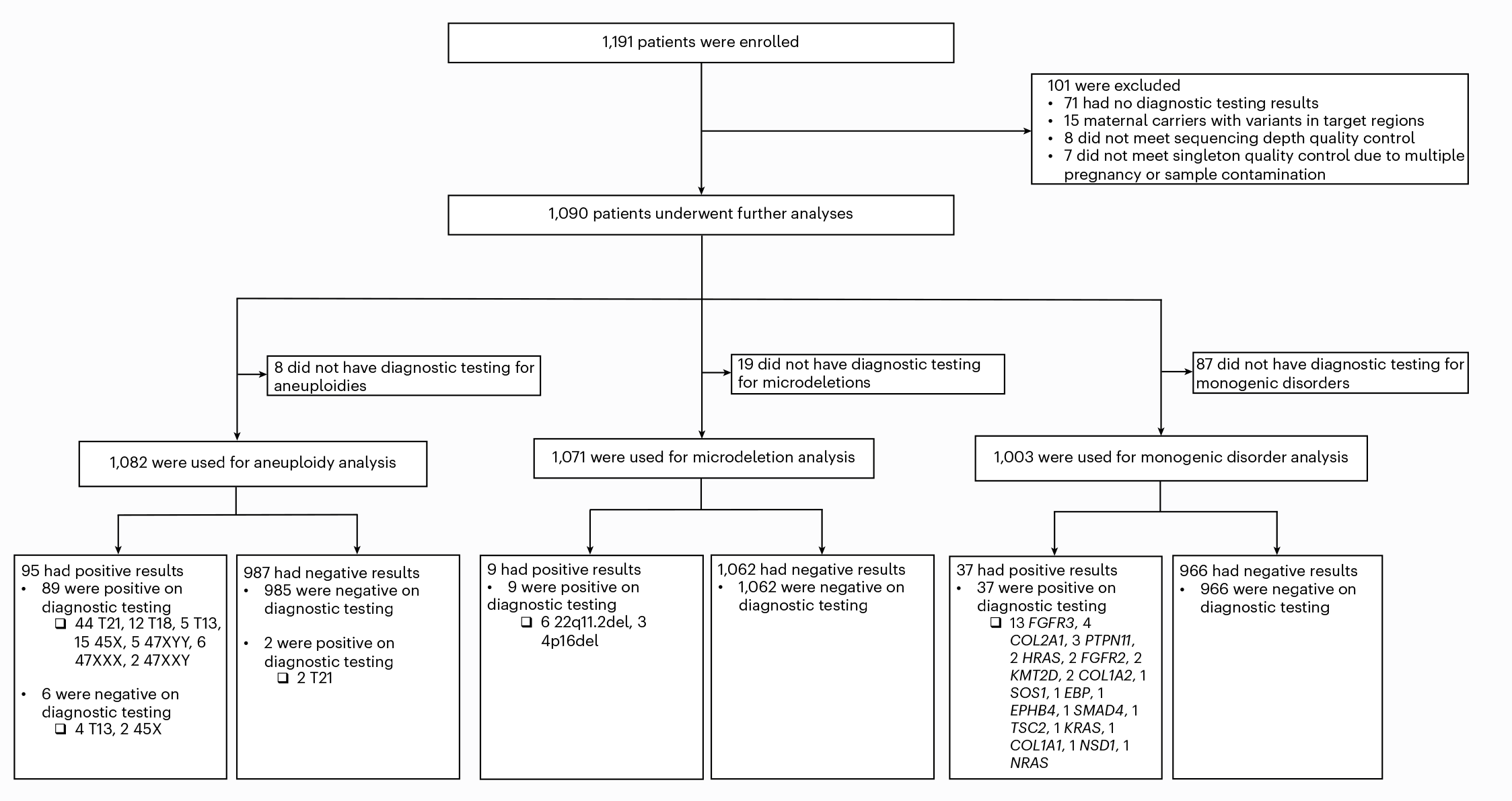
图 2. 针对多种遗传病的无创产前 cfDNA 综合性筛查的前瞻性、多中心临床研究
总体而言,cfDNA 三合一筛查对所有筛查疾病的临床检测灵敏度为 98.5%,特异性为 99.3% (表1.)。这些数值是将筛查结果与金标准的诊断结果比较后得出的。阳性预测值 (PPV) 和阴性预测值 (NPV) 分别为 95.7% 和 99.8% 。对于非整倍体、微缺失和单基因病,检测敏感性分别为 97.8%、100% 和 100%,特异性分别为 99.4%、100% 和 100% (表 1.) 。非整倍体、微缺失和单基因病受试者工作特征曲线下面积 (AUC) 分别为 0.996、1.000 和 1.000
(表 1.)。
表 1. 产前 cfDNA 三合一综合性筛查的临床有效性

在 cfDNA 三合一筛查中有 6 例假阳性病例,其诊断结果均为阴性。使用低深度全基因组测序的标准 cfDNA 的筛查方法,这些病例的筛查结果均为阳性。此外, cfDNA 三合一筛查还有两例 T21 的假阴性病例 (诊断测试结果为阳性) ,在标准 cfDNA 筛查中结果也为阴性。由于两种不同方法都对上述 8 例病例得出了错误的结果,它们不太可能由 cfDNA 筛查中的分析误差造成,而可能是由胎盘和胎儿之间的遗传差异造成的。已知限制性胎盘嵌合和胎儿限制性变异是导致产前 cfDNA 筛查和诊断结果不一致的因素。虽然 cfDNA 三合一筛查有 8 例错误的染色体结果,但在该研究中筛查的所有 37 例阳性和 966 例阴性病例中没有任何假阴性结果 (表 1.和图 2.)。
3.2 产前 cfDNA 三合一综合性筛查对胎儿结构异常的检出率
在超声筛查检测到结构异常的胎儿 (876 例) 中,98 例 (11.2%) 诊断出基因变异。其中,42例 (42.9%) 为常见的常染色体非整倍体 (T21、T18 和 T13) ,13 例 (13.3%) 为性染色体非整倍体,6 例 (6.1%) 为微缺失,37 例 (37.8%) 为单基因病 (图 3.
a)。基因变异诊断的检出率在淋巴系统异常中最高 (36.9%),其次是骨骼异常 (24.7%) 和多系统异常 (23.3%,图 3. b)。基因变异诊断的检出率随胎儿表型和潜在遗传病因的不同而明显不同。例如,32.6% 的淋巴系统异常的胎儿检出染色体异常,而仅有 4.3% 的病例是由单基因病引起的 (图 3. b)。总之,对于存在胎儿结构异常的孕妇,将目标单基因病与染色体异常结合同时分析,基因变异诊断的检出率增加了 60.7% (从 61 到 98,图 3. a)。
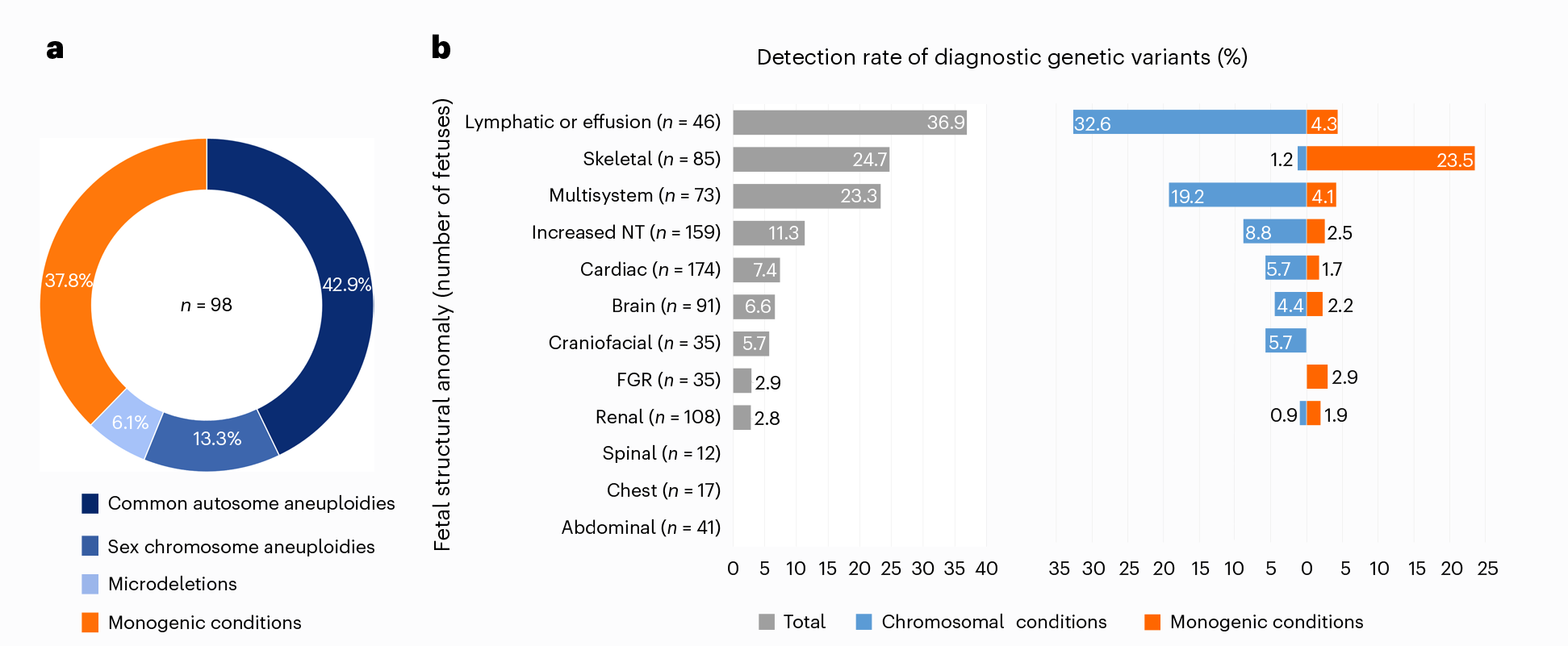
图 3. 产前 cfDNA 三合一综合性筛查在胎儿结构异常妊娠中对遗传变异诊断的检出率
3.3 产前 cfDNA 三合一综合性筛查参与者的妊娠结局
妊娠结局数据追踪至产后 6 周。对于最终队列的 1,090 名参与者,均进行了综合性产前 cfDNA 筛查和诊断检测。其中 623 例 (57.2%) 活产,268 例 (24.6%) 选择流产,2 例 (0.2%) 自然流产;另有 197 例 (18.1%) 没有妊娠结局数据。在诊断结果为阳性的 137 例中,有 11 例 (8.0%) 活产,106 例 (77.4%) 选择流产,1 例 (0.7%) 自然流产,19 例 (13.9%) 妊娠结局不明。对于经超声检查发现胎儿异常的 100 例,其中 4 例 (4.0%) 活产,82 例 (82.0%) 选择流产,13 例 (13.0%) 妊娠结局不明。对妊娠结局和所有产后和/或产前临床检查进行了评估,未发现遗传诊断与临床检查之间存在差异。所有临床妊娠管理决策均基于诊断结果。在所有提供了妊娠结局数据的案例中,未发现与进行 cfDNA 筛查或诊断相关的不良事件。
04 总结
该项研究强调了全面的胎儿 cfDNA 分析在增强严重遗传性疾病产前诊断方面的潜力。COATE-seq 技术与国内常用的低深度全基因组测序方法不同,通过有效降低等位基因杂交的偏好等技术创新,可更精准地检测胎儿 cfDNA 单核苷酸多态性 (SNP) 的等位基因频率。
这项多中心、前瞻性、观察性队列研究通过比较 cfDNA 筛查和诊断性检测结果,支持了包括人类遗传病三种最常见的病因——非整倍体、微缺失和单基因变异——在内的产前 cfDNA 综合筛查的临床有效性。该项研究凸显了胎儿 cfDNA 综合筛查在严重遗传病产前诊断方面的潜力,可以作为妊娠期早期识别的辅助工具,补充目前基于影像学的方案,有望填补产前筛查在显性单基因病方面的空白,促进知情的产前干预、围产期管理,并帮助父母的生殖决策。
参考文献
[1] Congenital disorders. WHO
https://www.who.int/news-room/fact-sheets/detail/birth-defects (2023).
[2] Mai C T, Isenburg J L, Canfield M A, et
al. National population‐based estimates for major birth defects, 2010–2014[J].
Birth defects research, 2019, 111(18): 1420-1435.
[3] Moore K L, Persaud T V N, Torchia M G.
The developing human-e-book: clinically oriented embryology[M]. Elsevier Health
Sciences, 2018.
[4] Xu C, Li J, Chen S, et al. Genetic
deconvolution of fetal and maternal cell-free DNA in maternal plasma enables
next-generation non-invasive prenatal screening[J]. Cell Discovery, 2022, 8(1):
109.