乳糖不耐人群新福音?羊奶的秘密被 298 头山羊“写进了基因”
新知小课堂
Q 为什么越来越多人选择羊奶?
山羊奶是一种营养丰富、易消化的天然乳品,脂肪球更小、含有天然益生元和 A2 型蛋白,对乳糖不耐和肠胃敏感人群更加友好;同时,它富含钙和维生素 D,有助于骨骼健康发育;相较奶牛,奶山羊养殖对环境资源的消耗更低,是可持续发展的优质奶源之一。初次尝试建议选择巴氏杀菌产品以减少腥膻味。
01 背景
萨能奶山羊 (Saanen dairy goats) 是世界范围内广泛养殖的高产乳用山羊品种,已在 80 多个国家形成了规模化饲养体系,其乳品在在发展中国家具有显著的经济效益。然而,尽管该品种在表型性状、产奶能力和遗传潜力方面表现出众,全球公开数据库中却仅收录了 5 个个体基因组数据,严重限制了分子育种和精准改良的进展。
近期,浙江大学联合西北农林科技大学在 Scientific Data (IF=5.8) 期刊上发表了题为“Whole-genome variants resource of 298 Saanen dairy goats”的研究性论文[1]。该研究依托 NadPrep® 高效建库技术对 298 头萨能奶山羊进行全基因组测序,构建了目前全球最大规模的萨能奶山羊基因组变异数据库,共鉴定出 1,460 万个 SNP 和 135 万个 InDel。这些数据填补了山羊基因组数据库的关键空白,为深入挖掘与产奶、抗病等重要性状相关的遗传标记提供了坚实基础,也为加速山羊品种改良与羊奶产业发展注入了蓬勃力量。该研究中运用 NadPrep® SP Beads 进行片段筛选和纯化,并采用 NadPrep® DNA Library Preparation Kit 完成文库构建,为高质量变异检测提供了关键技术保障。
02 研究方法
2.1 样本采集
选取来自浙江与陕西两地规模化养殖场共 298 头健康、无亲缘关系的萨能奶山羊母羊 (2 - 3 岁),每头采集 3 mL 血样。
2.2 DNA 提取、文库制备与基因测序
血液样本经标准化流程提取基因组 DNA 后进行超声片段化处理。随后,使用 NadPrep® SP Beads 进行片段筛选与纯化,并采用 NadPrep® DNA Library Prep Kit 完成预文库构建。最终在 DNBSEQ-T7 高通量测序平台上开展基因组测序。
2.3 数据分析
测序原始数据首先通过 fastp (v0.23.4) 进行质量控制与过滤,保留高质量序列。随后使用Burrows-Wheeler Aligner (BWA,v0.7.17) 将过滤后的序列比对至萨能奶山羊参考基因组 (GCA_042835985.1),并通过 SAM tools 完成排序、Picard 标记重复序列。基于 GATK (v4.1.5) 标准流程进行全基因组变异检测,识别 SNP 和 Indel 位点。在严格质控基础上,共获得 14.59 Mb 高质量 SNP 和1.34 Mb 高质量 InDel。所有变异位点进一步通过 VCFtools (v0.1.17) 进行缺失率控制 (<20%),最终构建完成萨能奶山羊的全基因组变异图谱。
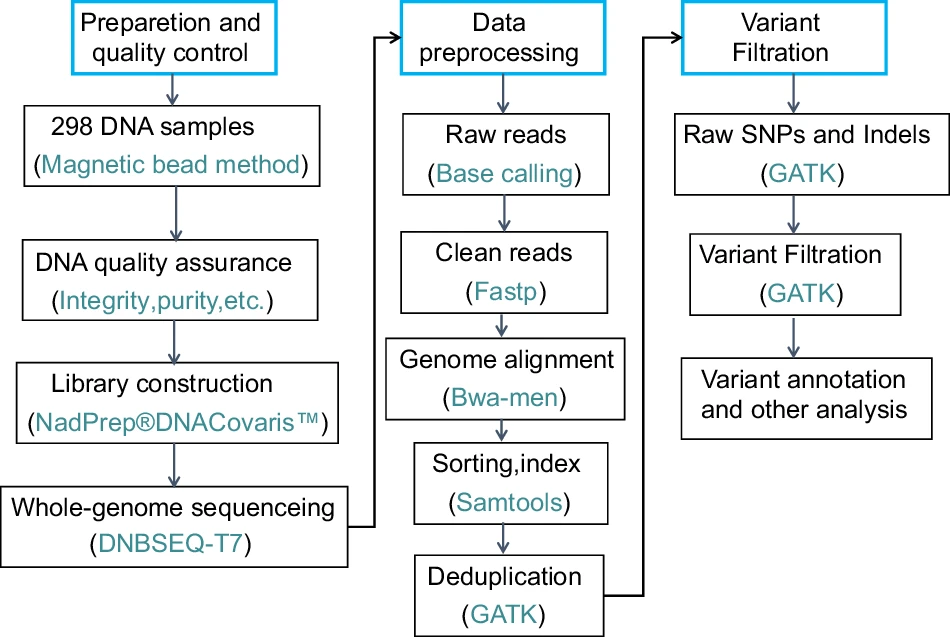
图 1. 样品质控、数据处理与变异筛选工作流程。该流程与 GATK 推荐的变体调用方案一致。
03 研究结果
3.1 测序数据质控
在测序数据质控环节,研究团队采用多维度评估体系,全面保障数据的准确性与可靠性。结果显示,单个样本平均产出 41.18 Gb 原始数据,经严格过滤后保留 41.07 Gb 高质量数据,平均有效数据率高达 99.82%。质量评估显示,92.3% 的碱基质量达到 Q30 标准,平均测序深度为 14.64 x,基因组比对率高达 99.9% (表 1.)。此外,通过箱线图 (图 2.) 直观展示了各样本测序深度、原始数据量及 Q30 比例的分布一致性,所有样本的质控指标均达到国际公认的群体基因组学研究标准,为后续变异识别和深入分析提供了可靠的数据基础。
表 1. 测序数据汇总表。表中所示为所有样本指标均值。
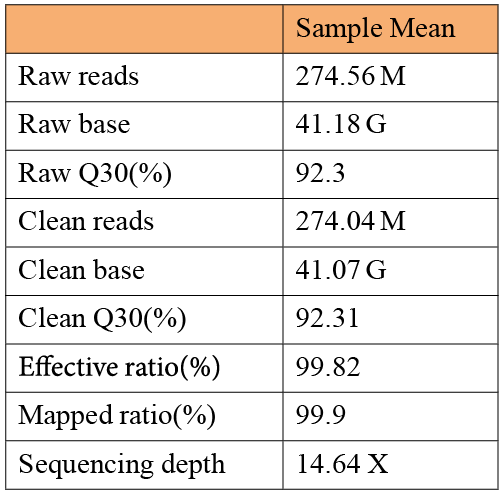
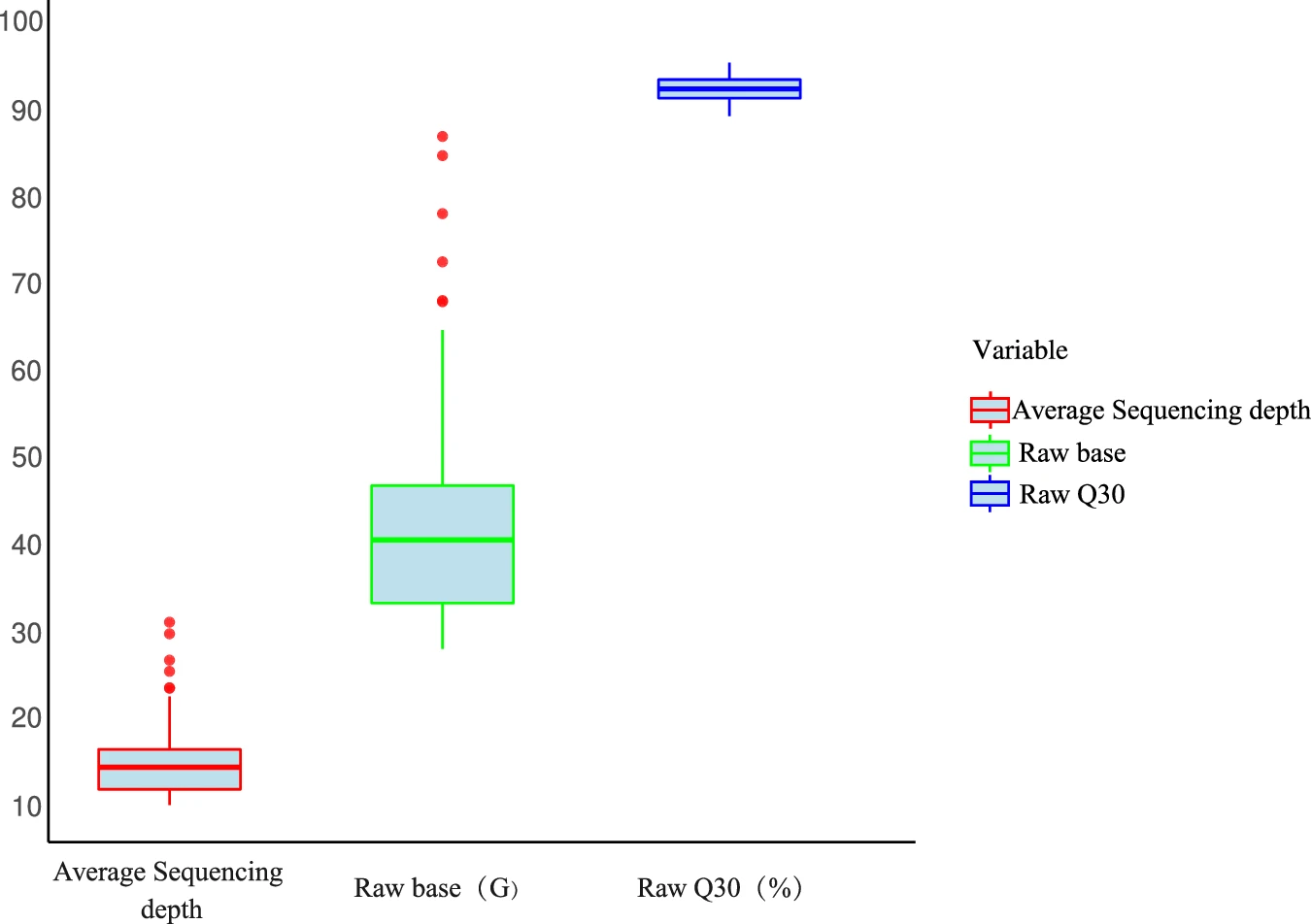
图 2. 样品的平均测序深度、原始碱基和原始 Q30 (n = 298)。
3.2 SNP 和 InDel 数据质控
该研究采用统一分析流程对山羊群体进行全基因组变异检测,初步识别出 2,980 万个 SNP 和 349 万个 InDel。随后,通过 GATK 的 Variant Filtration 模块,结合比对质量 (MQ)、深度质量比 (QD)、链偏差检验 (FS) 和链偏好比值 (SOR) 等多维度参数进行严格质控,最终获得 14,597,388 个高质量 SNP 和 1,348,195 个高质量 InDel。从基因组分布特征来看,平均每 1 kb区域含有 6 个 SNP,每 2 kb 区域含有 1 个 InDel (图 3.a-b,表 2.)。功能注释结果显示,SNP 位点主要分布于基因间区 (78.3%)、内含子区 (19.4%) 和外显子区 (0.7%);InDel 的对应分布比例为 77.6%、20.2% 和 0.2% (表 3. & 图 4. a-b)。综合质控结果表明,该研究从测序、比对到变异筛选的全流程分析方法具有高度科学性与稳定性。所构建的变异数据集将为后续群体遗传学、分子育种与功能基因研究提供重要支持。
表 2. 各染色体中 SNP 和 InDel 的统计汇总。变异密度按每千碱基 (Kb) 长度内的突变数计算。
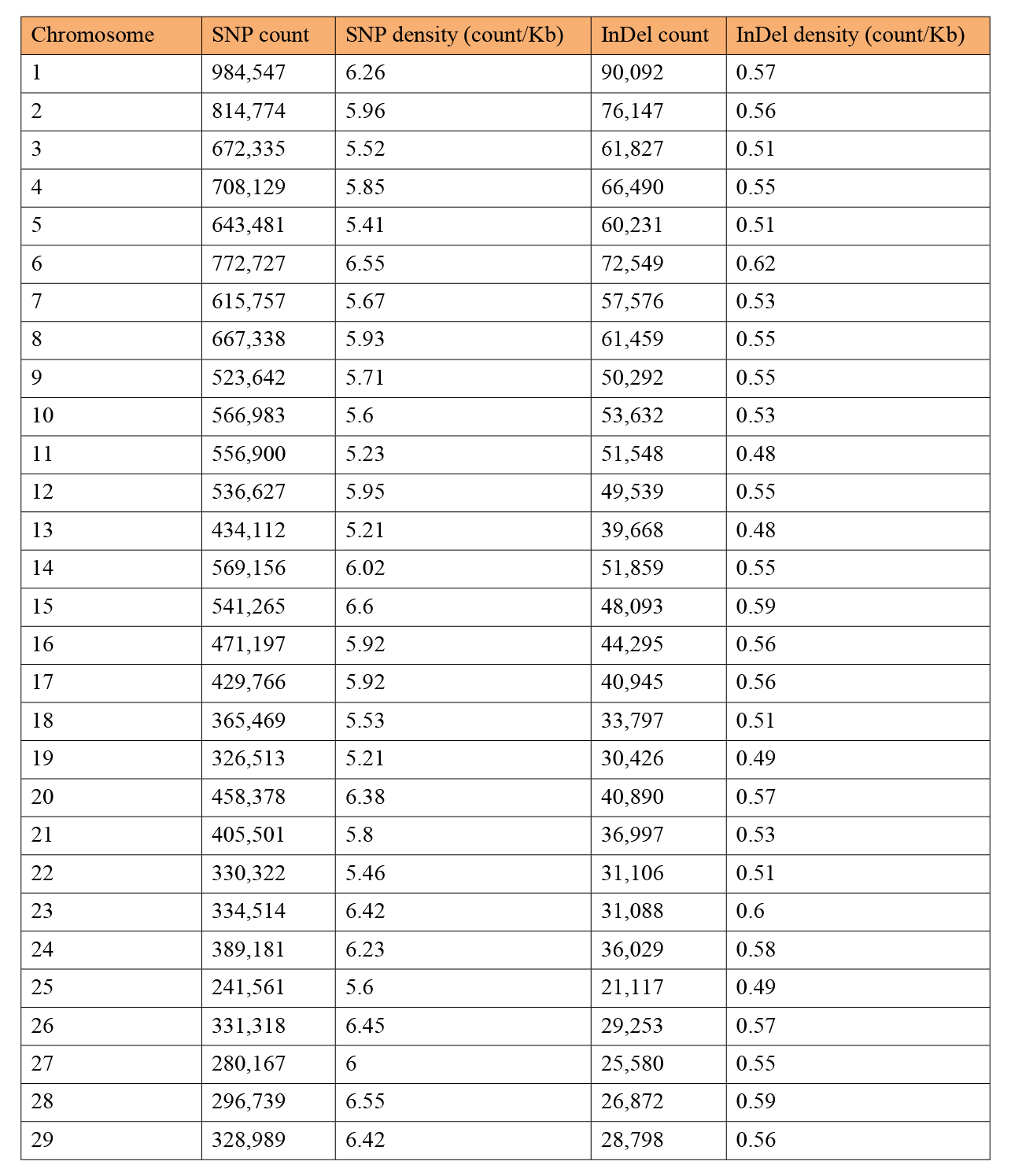
表 3. SNP 和 InDel 在不同功能注释类别中的分布情况。
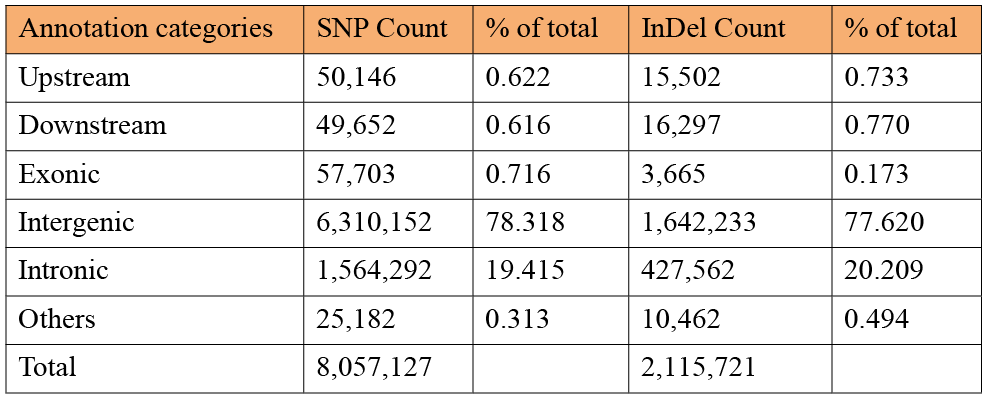
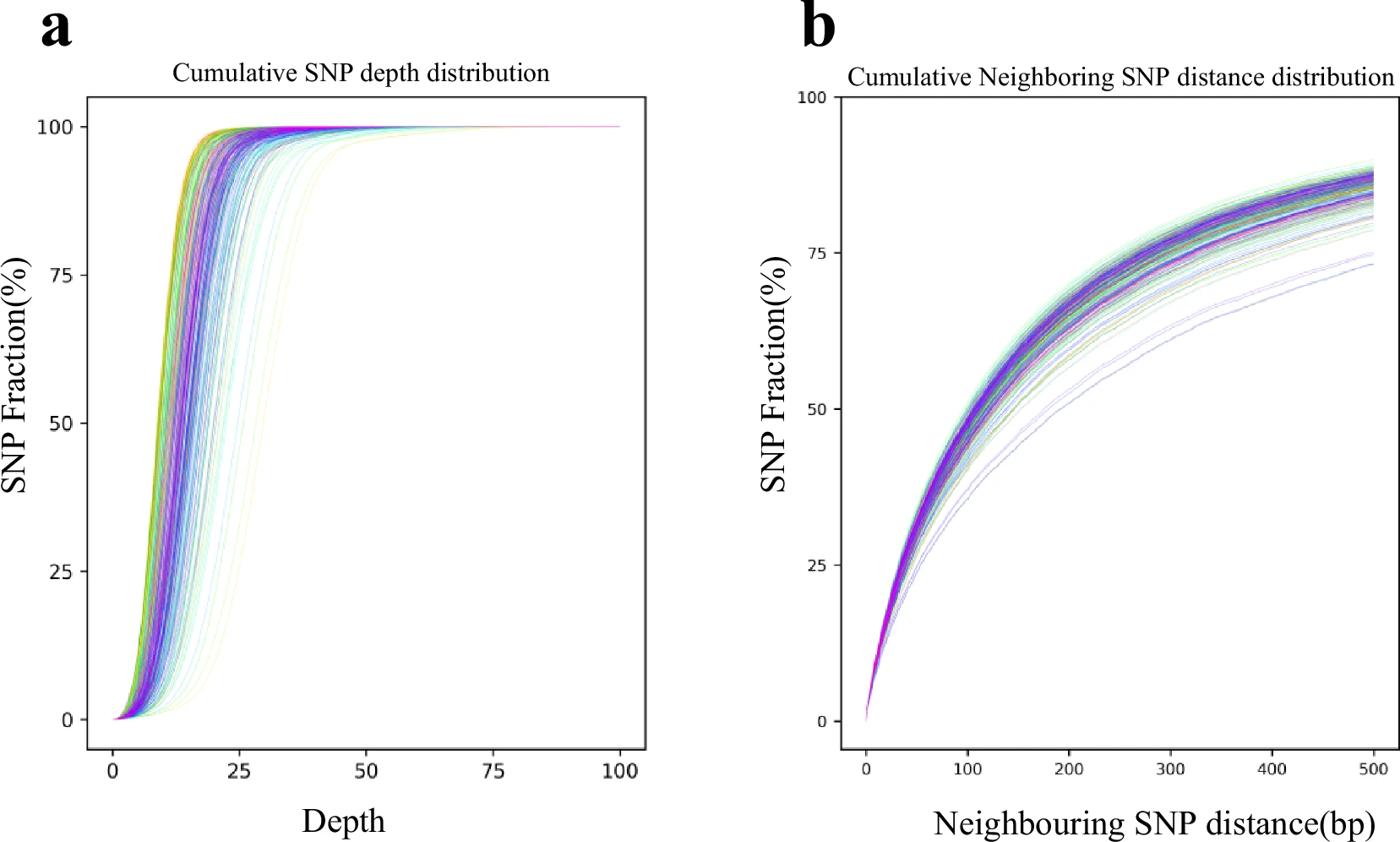
图 3. SNP 深度分布统计 (a) 及相邻 SNP间距分布统计 (b)。不同颜色代表不同个体样本。
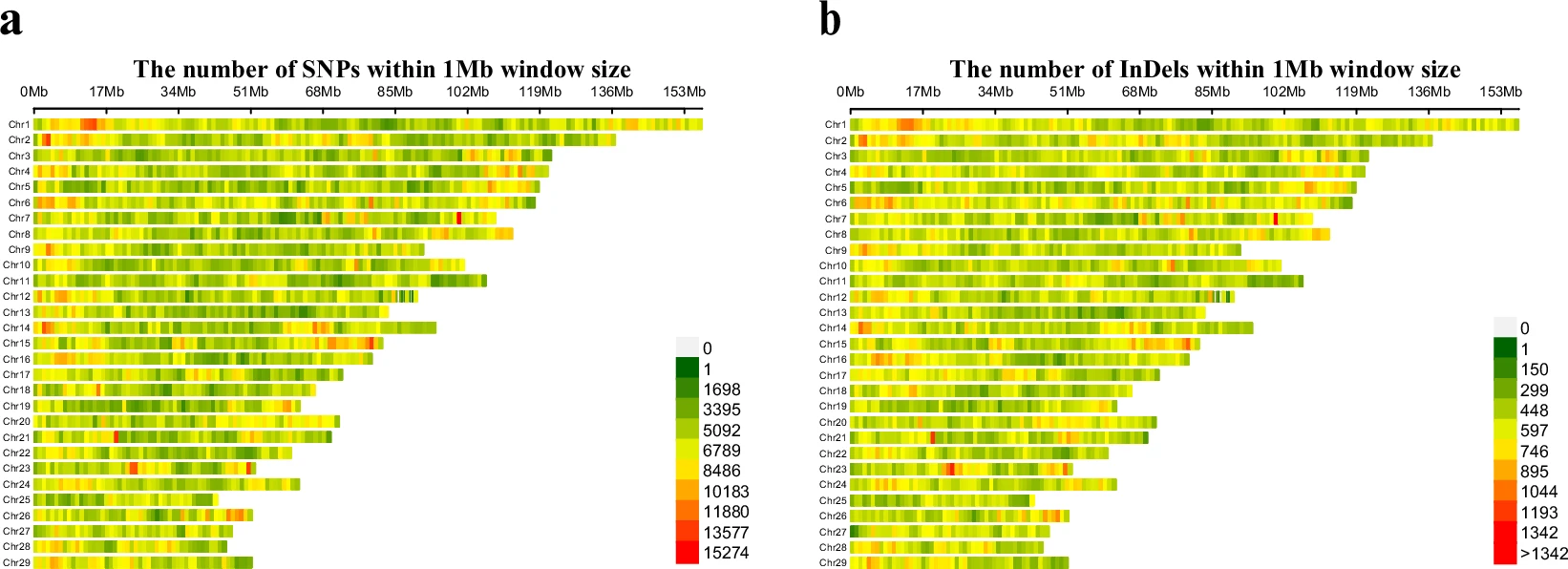
图 4. 298 头中国萨能奶山羊全基因组范围内 SNP 和 InDel 的分布情况。a. 全基因组范围内的 SNP密度统计,颜色梯度反映每 1 Mb 窗口内 SNP 数量的变化;b. 全基因组范围内的 SNP 和 InDel 密度统计。
注:X 轴表示染色体位置,Y 轴标注染色体编号,不同色块对应不同密度区间。
04 总结与展望
该研究基于全基因组测序数据,结合 GATK 标准分析流程,系统鉴定了中国萨能奶山羊全基因组范围内的 SNP 和 InDel (MAF≥0.5%),构建了高质量的遗传变异数据集。尽管本研究尚未涵盖性染色体与结构变异的相关分析,但已为群体遗传学研究、分子育种以及重要经济性状的遗传机制研究奠定了坚实基础。未来,随着更多功能注释和表型数据的整合,可进一步深入挖掘这些变异类型在性状形成中的作用,识别影响表型多样性与疾病易感性的重要基因组位点,推动精准育种与功能基因研究的深入发展。
参考文献
[1] Zhang K, Zhao J, Mi S, et al. Whole-genome variants resource of 298 Saanen dairy goats[J]. Scientific Data, 2025, 12(1): 528.