从“形态主导”到“基因定义”,血液肿瘤检测的“金标准”定了?
01 背景概述
传统上,血液肿瘤的分子遗传学改变主要通过常规细胞遗传学、荧光原位杂交或 PCR 技术检测。相较于单次检测的逐级分步策略,NGS 在多样本中同步检测多重异常的能力显著提升了成本效益并缩短了检测周期。基于这一技术优势,NGS 已成为解析血液肿瘤分子特征的核心手段。
02 靶向 NGS:DNA Panel
基于 DNA 的捕获测序是目前血液肿瘤分子特征分析最广泛使用的选择。
在世界卫生组织第 5 版《造血与淋巴组织肿瘤分类》 (WHO2022) 中,进一步扩展了由遗传学异常定义的疾病亚型,使得靶向 NGS 检测的重要性更加凸显。克隆性造血 (CH) 指健康个体多能干细胞/祖细胞中体细胞突变的累积[1]。WHO2022 新增了髓系前驱病变类别,包括意义未明的克隆性造血 (CHIP) 和意义未定的克隆性血细胞减少 (CCUS)。CHIP 特指未确诊血液疾病或血细胞减少的个体中,携带髓系恶性肿瘤相关基因体细胞突变且变异等位基因频率 (VAF) ≥ 2% 的 CH。当 CHIP 合并无法解释的持续性血细胞减少时,则定义为 CCUS。常见 CH 驱动突变涉及 DNMT3A、TET2、ASXL1、JAK2、TP53、SF3B1、PPM1D、SRSF2、ZBTB33、IDH1、IDH2、U2AF1、KRAS、NRAS、CTCF、CBL、GNB1、BRCC3、PTPN11、GNAS、BCOR 及 BCORL1 等基因[2]。
WHO2022 将伴 SF3B1 突变的骨髓增生异常综合征 (MDS) 列为“具有明确遗传学异常的 MDS”亚类之一。该分类通过识别具有高风险特征和不良预后的特定类别,强调了多重 TP53 改变的重要性。最新发布的《MDS 分子国际预后评分系统》中,不良生物标志物包括多重 TP53 改变、FLT3 突变及 KMT2A 部分串联重复,而 SF3B1 突变则与良好预后相关[3]。具有显著预后价值的 16 个基因为 TP53、KMT2A、FLT3、SF3B1、NPM1、RUNX1、NRAS、ETV6、IDH2、CBL、EZH2、U2AF1、SRSF2、DNMT3A、ASXL1 和 KRAS。
WHO2022 扩展了定义特定急性髓系白血病 (AML) 亚群的基因突变谱。NPM1 和 CEBPA 仍为 AML 定义性突变。值得注意的是,WHO2022 将 CEBPA 突变 AML 限定为具有双等位 CEBPA 突变或单等位基因内 bZIP 区框内突变。这反映了 CEBPA 基因 bZIP 区框内突变具有独特的临床与分子特征,包括发病年龄较轻、GATA2 和 NPM1 共突变频率高、治疗反应佳且生存改善[4, 5]。新版分类摒弃了“伴骨髓增生异常相关改变的 AML”这一术语,转而引入与继发性肿瘤特征及不良预后相关的细胞遗传学与分子异常。WHO2022 将该类 AML 定义为“骨髓增生异常相关 AML (AML-MR)”,并列出 8 个诊断相关基因 (ASXL1、BCOR、EZH2、SF3B1、SRSF2、STAG2、U2AF1 和 ZRSR2)。
分子分析技术的进步揭示,髓系肿瘤患者存在胚系易感基因的比例远超既往认知[6]。这对临床实践具有重要影响,包括异基因造血干细胞移植供体选择、预处理方案制定、家族成员筛查及遗传咨询等。WHO2022 新增“继发性髓系肿瘤”类别,涵盖 CEBPA、DDX41、TP53、RUNX1、ANKRD26、ETV6 等胚系致病/可能致病突变,以及器官功能障碍综合征相关突变 (如范可尼贫血、唐氏综合征、RAS 病等)。其中,DDX41 胚系突变值得特别关注,这是 AML 和 MDS 中最常见的遗传易感突变。研究表明,AML 的发生与同一基因在胚系突变背景下发生二次突变相关。伴 DDX41 胚系突变的 AML 具有男性多发、中位发病年龄 70 岁、白细胞计数低、化疗反应佳及总体预后良好等特征[7, 8]。
更新的《2022 欧洲白血病协作网 (ELN2022) 成人 AML 风险分层》整合了最新分子发现与临床试验证据[9]。相较于既往指南,ELN2022 的主要更新包括:第一,FLT3 内部串联重复 (ITD) 等位基因比 (AR) 不再纳入风险分层,因此所有 FLT3-ITD 突变 AML 患者均归为中危组;第二,基于 WHO2022 定义的 AML-MR 相关基因突变患者现归类为预后不良组,RUNX1 突变虽未被 WHO2022 列为 MR 标准,但在 ELN2022 中仍视为不良遗传学异常;第三, CEBPA 突变 AML 的良好预后仅与影响 bZIP 区的框内突变相关;第四,VAF >
10% 的 TP53 突变被视为高危组。除诊断与风险分层外,识别靶向治疗相关分子标志物对临床决策具有直接指导意义。目前 FDA 批准的 AML 靶向治疗相关基因包括 FLT3-ITD、酪氨酸激酶结构域突变及 IDH1/IDH2 突变[10, 11]。
相较于髓系肿瘤,淋巴系统肿瘤诊断对基因突变的依赖度较低。WHO2022 将 PAX5 突变纳入“伴其他明确遗传学异常的B急性淋巴细胞白血病 (ALL)”新亚型。部分基因突变已被用于支持特定淋巴瘤诊断,如毛细胞白血病的 BRAF 突变、淋巴浆细胞淋巴瘤的 MYD88 或 CXCR4 突变、EBV 阴性 Burkitt 淋巴瘤的 TCF3 或 ID3 突变、T 大颗粒淋巴细胞白血病的 STAT3 或 STAT5B 突变等。浆细胞骨髓瘤 (PCM) 中,KRAS、NRAS、IRF4、MAX、HIST1H1E、RB1、EGR1、TP53、TRAF3、FAM46C、DIS3、BRAF、LTB、CYLD 及 FGFR3 等基因呈现高频复发突变[12, 13]。
03 靶向 NGS:RNA Panel
在临床血液学检测中,基于 RNA 的捕获测序主要用于分析融合转录本以及断点。
尽管 WHO2022 保留了多数 AML 定义性基因融合,但“伴明确遗传学异常的 AML”相关融合基因分类仍有重要更新:第一,伴 BCR::ABL1 融合的 AML 被列为正式亚型;第二,“伴 t(9;11)(p22;q23);KMT2A-MLLT3 的 AML”更名为“伴 KMT2A 重排的 AML”,因已发现超过 100 种 KMT2A 融合伴侣[14]。尽管融合伴侣鉴定非必需,但其可提供预后信息并指导疗效监测;第三,“伴 NUP98 重排的 AML”被列为新亚型,该基因多与隐性伙伴发生重排,通常提示不良预后[15, 16]。
ALL 的融合基因分类基本延续既往标准,主要更新体现在新增融合基因类型及基于基因表达特征的亚型优化:第一,新增罕见亚型“伴 TCF3::HLF 融合的 B-ALL”,该亚型预后极差;第二,“BCR::ABL1 样 B-ALL”成为正式分类,其特征为基因表达谱 (GEP) 聚类与 BCR::ABL1 阳性 B-ALL 相似但缺乏该融合。由于分子异常多样性及对 GEP 芯片技术的依赖,此类诊断具有挑战性。靶向 RNA 测序可作为替代诊断工具,因该亚型常伴 CRLF2、JAK2、ABL1、PDGFRB、ABL2、EPOR、PTK2B、CSF1R、DGKH、IL2RB、NTRK3、TSLP 及 TYK2 等基因融合;第三,“伴其他明确遗传学异常的 B-ALL”包括 MYC、DUX4、MEF2D、ZNF384 或 NUTM1 重排以及 PAX5 改变。目前 T-ALL 尚缺乏足够证据建立遗传学定义亚型。PCM 中,IGH 基因是主要融合伙伴,少数涉及 MYC。
表 1. 血液肿瘤中具有明确临床意义的突变基因。
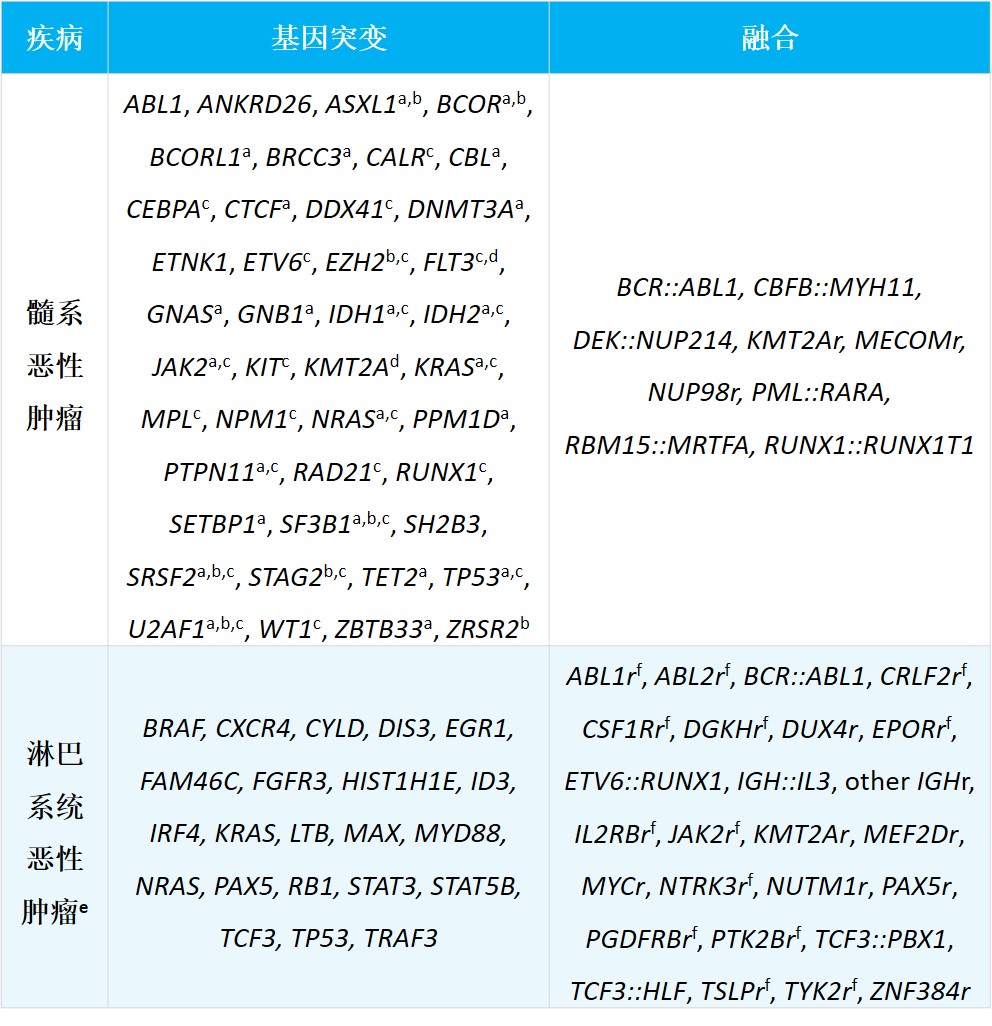
a 克隆造血中常见突变的基因[2]
b 根据 WHO2022 分类,这些基因突变的存在定义了“急性髓细胞白血病骨髓增生异常相关”类别[2]
c ELN 指南提供的基本基因集可能有助于急性髓系白血病可测量的残留疾病监测的小组方法[17]
d 使用靶向 NGS 检测 FLT3 内部串联重复或 KMT2A 部分串联重复的可靠检测可能需要专门的生物信息学分析
e 仅考虑血液或骨髓源性淋巴系统恶性肿瘤的遗传异常
f 基因通常参与表征 BCR:ABL1 样特征的基因融合
04 靶向之外的 NGS
一些研究采用 WGS/WTS 进行血液恶性肿瘤基因组图谱分析。WGS 可检出所有细胞遗传学分析识别的复发易位与拷贝数变异,前瞻性测序为 25% 患者提供新遗传学信息,部分患者因此改变风险分层[18]。儿童 ALL 全基因组研究显示,WGS 几乎可检测所有亚型定义性遗传学异常,并发现 MAP 激酶通路基因融合等新型变异[19]。AML/MDS 的 WTS 研究发现,37% AML 和 3% MDS 患者存在融合基因,其中 AML 患者 41% 的融合及 MDS 患者 88% 的融合为首次报道[20]。尽管 WGS/WTS 揭示了血液恶性肿瘤分子特征的复杂性,但其高昂成本及复杂生物信息学分析要求限制了临床常规应用。
05 结论
NGS 已深度融入血液恶性肿瘤全程管理的分子特征解析。其应用贯穿诊断、预后评估至治疗后监测。但市面多数 Panel 仅限于检测 DNA 或靶向已知热点,难以覆盖 WHO2022 新增基因与检测类型,亟需技术升级。纳昂达即将推出专为血液肿瘤设计的“DNA + RNA双维度”靶向测序方案——NanoHema Panel v2.0,以 481 基因全景覆盖、DNA-RNA 双维验证的设计,助力 NGS 在实践中的转化应用,敬请期待!
参考文献
[1] Jaiswal S, Fontanillas P, Flannick J, et al. Age-related clonal hematopoiesis associated with adverse outcomes[J]. New England Journal of Medicine, 2014, 371(26): 2488-2498.
[2] Khoury J D, Solary E, Abla O, et al. The 5th edition of the World Health Organization classification of haematolymphoid tumours: myeloid and histiocytic/dendritic neoplasms[J]. leukemia, 2022, 36(7): 1703-1719.
[3] Bernard E, Tuechler H, Greenberg P L, et al. Molecular international prognostic scoring system for myelodysplastic syndromes[J]. NEJM evidence, 2022, 1(7): EVIDoa2200008.
[4] Wakita S, Sakaguchi M, Oh I, et al. Prognostic impact of CEBPA bZIP domain mutation in acute myeloid leukemia[J]. Blood Advances, 2022, 6(1): 238-247.
[5] Taube F, Georgi J A, Kramer M, et al. CEBPA mutations in 4708 patients with acute myeloid leukemia: differential impact of bZIP and TAD mutations on outcome[J]. Blood, The Journal of the American Society of Hematology, 2022, 139(1): 87-103.
[6] Klco J M, Mullighan C G. Advances in germline predisposition to acute leukaemias and myeloid neoplasms[J]. Nature Reviews Cancer, 2021, 21(2): 122-137.
[7] Bataller A, Loghavi S, Gerstein Y, et al. Characteristics and clinical outcomes of patients with myeloid malignancies and DDX41 variants[J]. American journal of hematology, 2023, 98(11): 1780-1790.
[8] Duployez N, Largeaud L, Duchmann M, et al. Prognostic impact of DDX41 germline mutations in intensively treated acute myeloid leukemia patients: an ALFA-FILO study[J]. Blood, The Journal of the American Society of Hematology, 2022, 140(7): 756-768.
[9] Döhner H, Wei A H, Appelbaum F R, et al. Diagnosis and management of AML in adults: 2022 recommendations from an international expert panel on behalf of the ELN[J]. Blood, The Journal of the American Society of Hematology, 2022, 140(12): 1345-1377.
[10] Byun J M, Yoo S J, Kim H J, et al. IDH1/2 mutations in acute myeloid leukemia[J]. Blood research, 2022, 57(1): 13-19.
[11] Ahn J S, Kim H J. FLT3 mutations in acute myeloid leukemia: A review focusing on clinically applicable drugs[J]. Blood research, 2022, 57(S1): 32-36.
[12] Walker B A, Boyle E M, Wardell C P, et al. Mutational spectrum, copy number changes, and outcome: results of a sequencing study of patients with newly diagnosed myeloma[J]. Journal of clinical oncology, 2015, 33(33): 3911-3920.
[13] Manier S, Salem K Z, Park J, et al. Genomic complexity of multiple myeloma and its clinical implications[J]. Nature reviews Clinical oncology, 2017, 14(2): 100-113.
[14] Meyer C, Larghero P, Almeida Lopes B, et al. The KMT2A recombinome of acute leukemias in 2023[J]. Leukemia, 2023, 37(5): 988-1005.
[15] Xie W, Raess P W, Dunlap J, et al. Adult acute myeloid leukemia patients with NUP98 rearrangement have frequent cryptic translocations and unfavorable outcome[J]. Leukemia & Lymphoma, 2022, 63(8): 1907-1916.
[16] Bertrums E J M, Smith J L, Harmon L, et al. Comprehensive molecular and clinical characterization of NUP98 fusions in pediatric acute myeloid leukemia[J]. Haematologica, 2023, 108(8): 2044.
[17] Heuser M, Freeman S D, Ossenkoppele G J, et al. 2021 Update on MRD in acute myeloid leukemia: a consensus document from the European LeukemiaNet MRD Working Party[J]. Blood, The Journal of the American Society of Hematology, 2021, 138(26): 2753-2767.
[18] Duncavage E J, Schroeder M C, O’Laughlin M, et al. Genome sequencing as an alternative to cytogenetic analysis in myeloid cancers[J]. New England Journal of Medicine, 2021, 384(10): 924-935.
[19] Ryan S L, Peden J F, Kingsbury Z, et al. Whole genome sequencing provides comprehensive genetic testing in childhood B-cell acute lymphoblastic leukaemia[J]. Leukemia, 2023, 37(3): 518-528.
[20] Stengel A, Shahswar R, Haferlach T, et al. Whole transcriptome sequencing detects a large number of novel fusion transcripts in patients with AML and MDS[J]. Blood Advances, 2020, 4(21): 5393-5401.







