DNA 甲基化测序该如何选?EM or BS?
# 点击蓝字 关注我们 #
前言
DNA 的甲基化修饰会影响基因的表达,例如转录起始位点附近的低甲基化与较高的转录相关。目前,DNA 甲基化图谱分析在癌症等疾病研究、胚胎发育监测和农业植物研究中被愈加重视。大量研究表明,多种类型癌症中的 DNA 甲基化异常会导致大范围的基因沉默。在癌症早筛早检方面,ctDNA 的甲基化信号比基因突变的信息更加丰富,可进行组织溯源并识别组织分化状态。相比于全基因组甲基化测序,靶向甲基化测序在成本上表现更加优秀[1]。
DNA 甲基化检测首先要进行甲基转化,一种是重亚硫酸盐法(Bisulfite,BS),将未甲基化的 C 转化为 U。尽管这种方式是最常用和高效的方法,但因其高温强碱的反应条件会导致 DNA 断裂损伤,不适合应用在珍稀样品中,同时也会导致文库比对率和覆盖均一性的降低。另一种方式是利用较为温和的酶转化方式(Enzyme,EM),以减少 DNA 的损伤,但转化效率可能略低[2]。
本文中,我们主要分享纳昂达甲基化测序方案应用于标准品和真实样本的示例,并对比 BS 和 EM 转化方式,以及生信分析经验分享。
甲基化测序方案
纳昂达科技提供甲基化靶向富集解决方案,包括:基于重亚硫酸盐法或酶转化的甲基化测序文库构建试剂盒、针对转化后文库的液相杂交捕获探针设计及合成服务、液相杂交捕获体系及甲基化靶向富集 NGS 自动化分析平台:
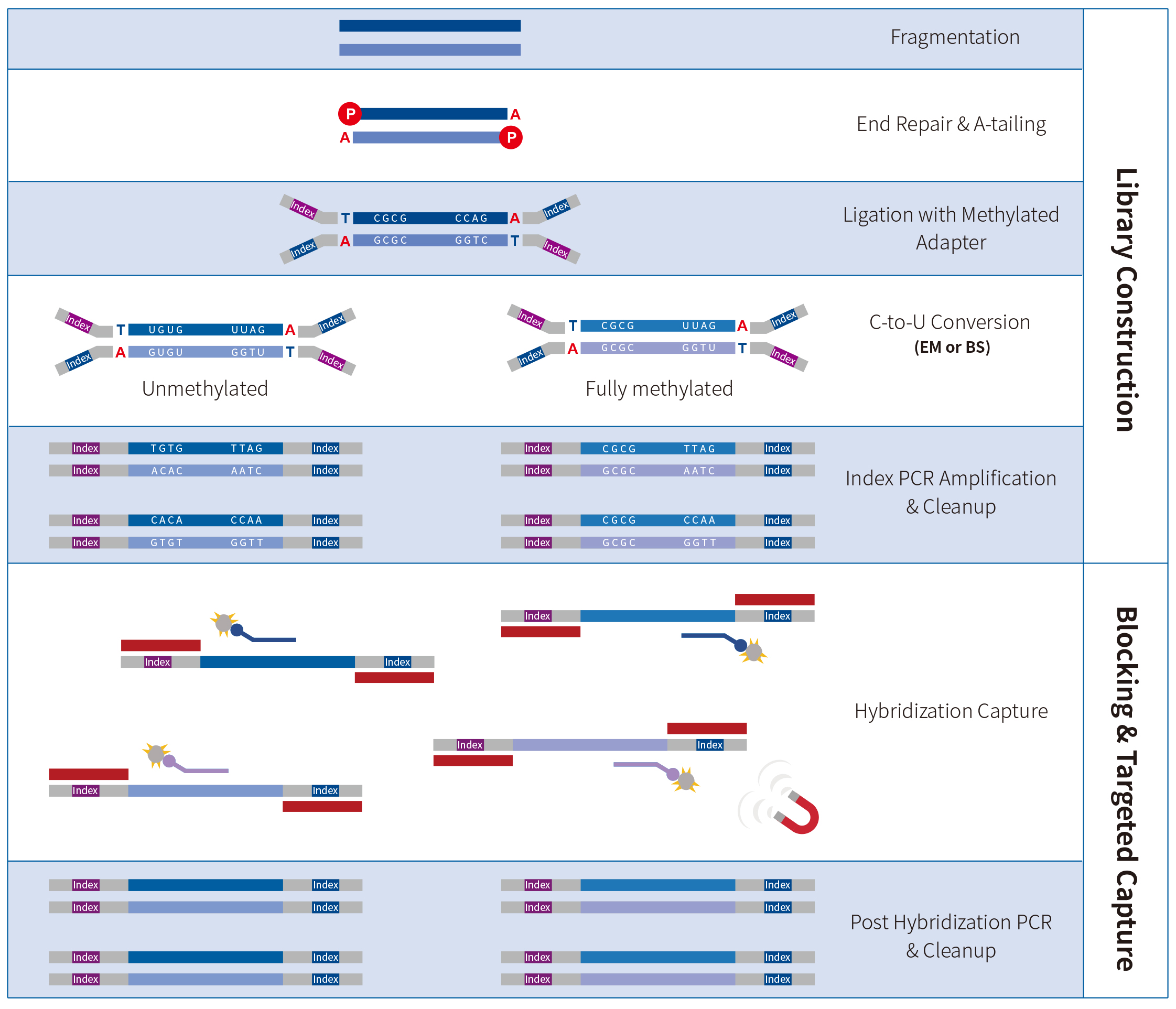
实验设计
样本:甲基化标准品为人甲基化阳性对照 100% Methylated DNA (ZYMO, D5014-2) 和阴性对照 0% Methylated DNA (ZYMO, D5014-1),经不同比例混合以模拟不同甲基化水平:0%,10%,50% 和 100%,投入量均为 50 ng。真实样本为匿名卵巢癌 FFPE 样本,投入量均为 100 ng。
靶向捕获 Panel:Meth-GC Panel 靶向胃癌相关基因启动子区域及 CpG 位点,共包含 5017 个 CpG 位点,捕获区域约为 60 kb(用于标准品);Meth-HR Panel 靶向 14 个 HR 通路的基因启动子区域,共包含 507 个 CpG 位点,捕获区域约为 5 kb(用于真实样本)。
其它相关试剂见下表。
表 1. 相关试剂

分析软件:Bismark[3]、BWA-meth[4] 和 Msuite[5] 软件应用于甲基化测序数据的比对,Msuite 和 Bismark 底层均采用 bowtie2 进行比对,而 BWA-meth 则采用 BWA 进行比对。Methlykit[6]、eDMR[7] 和DSS[8] 软件用于真实样本的差异甲基化区域(Differentially Methylated Region,DMR)分析。DSS 软件核心是一个基于贝叶斯层次模型的程序,用于估计 CpG 位点特异性分散,然后进行 Wald-tests 以检测 DMR;eDMR 是基于双峰正态分布模型和成本函数优化进行 DMR 分析;Methlykit 则是采用 Fisher 精确检验或逻辑回归检验来计算 DMR 的显著性。DSS 和 Methlykit 均可以对 DMP 和 DMR 进行分析,而 eDMR 只能对 DMR 进行分析。这三款软件均使用默认参数分析。
表 2. 分析软件列表

不同转化方式下的甲基化检测
纳昂达科技的甲基化文库构建试剂盒支持 BS 和 EM 两种转化方式。EM 转化方式较为温和,文库得量显著高于 BS 转化方式,相应的插入片段也更长一些(图1 A & C)。但是随着投入量的增加,EM 的转化效率会降低(图1 B)。

图 1. 不同转化方式的建库表现。A.文库产出,B. 不同投入量 λ DNA C 碱基转化效率及 C. 插入片段分布。
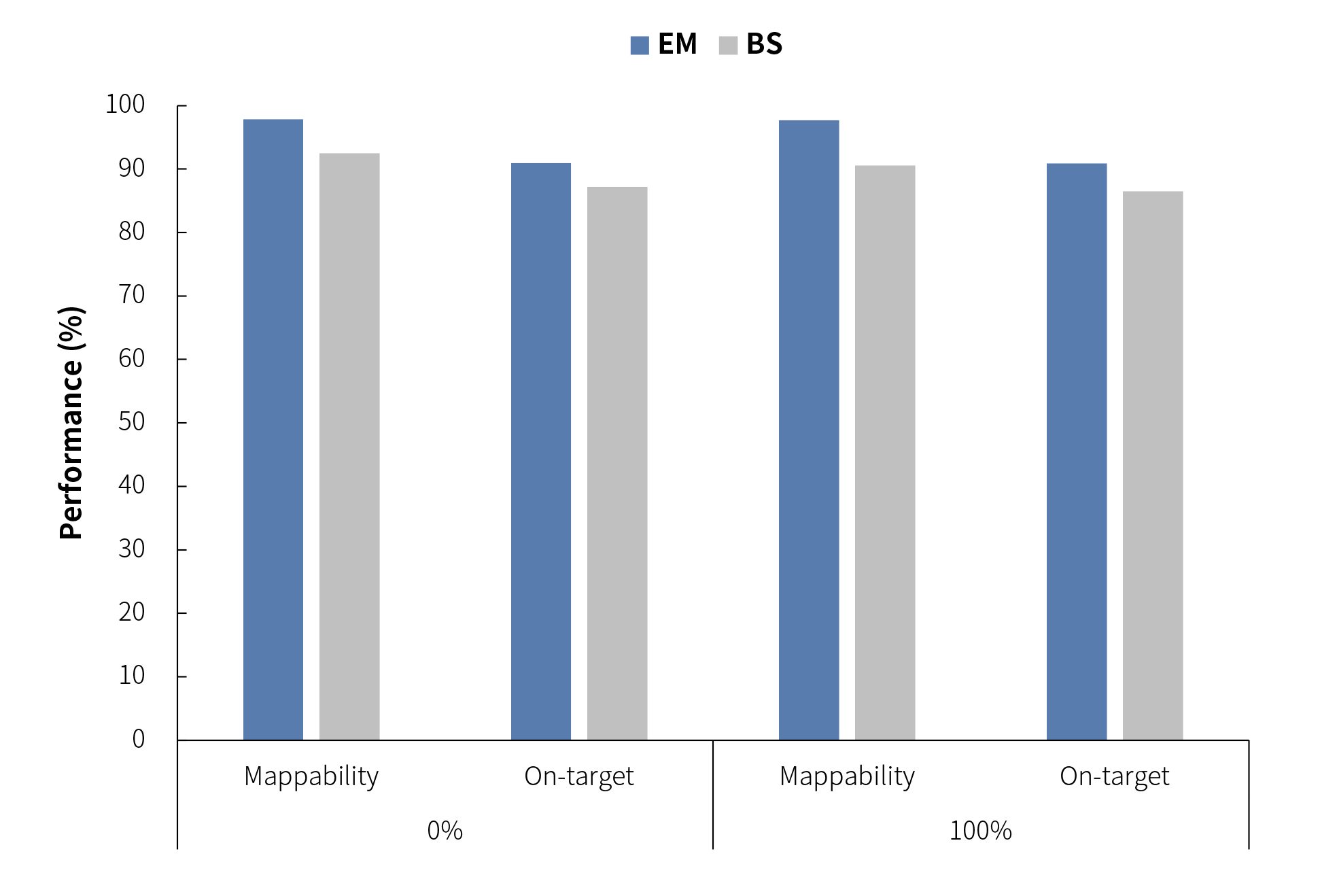
当使用 BWA-meth 比对时,Meth-GC Panel 靶向捕获的结果显示两种转化方式的中靶率均 > 85%,覆盖均一性上也均有优异的表现,但 BS 转化的有效数据量更低,比对率和中靶率均低于 EM 转化(图2)。
图 2. 标准品靶向甲基化捕获结果
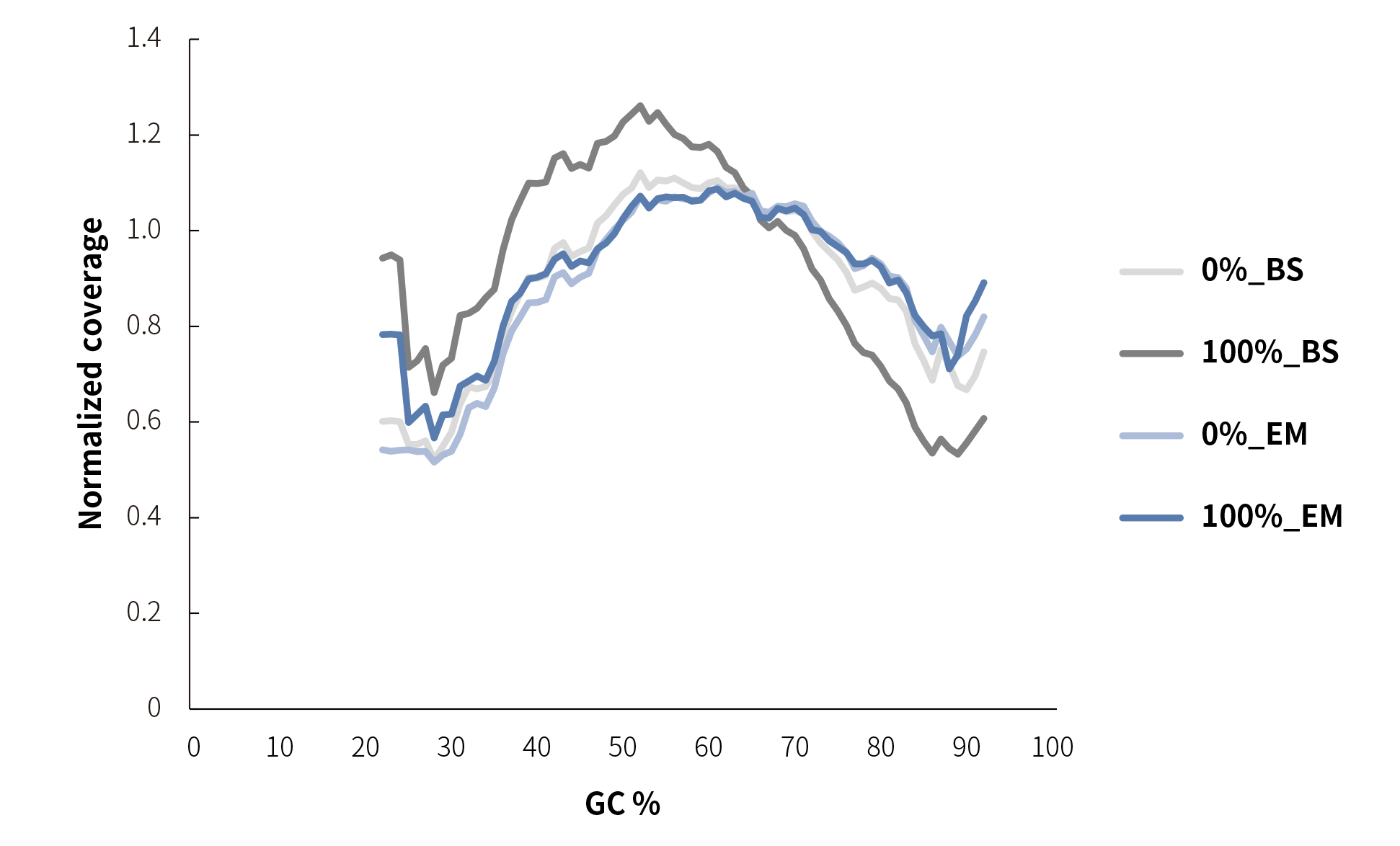
考虑到 BWA-meth 软件本身针对高 GC 区域进行了优化,我们也与两款调用 bowtie2 比对的软件 Bismark 和 Msuite 进行了对比。5017 个 CpG 位点深度的相关性热图显示,BWA-meth 和 Bismark 的相关性很高,均 > 0.7;而 Msuite 跟前两者的相关性很较差(图3),CpG 位点深度偏低,主要原因很可能是 Msuite 软件对读长筛选和质量过滤跟前两者存在较大差异。

图 3. 标准品 CpG 位点不同软件检测覆盖结果
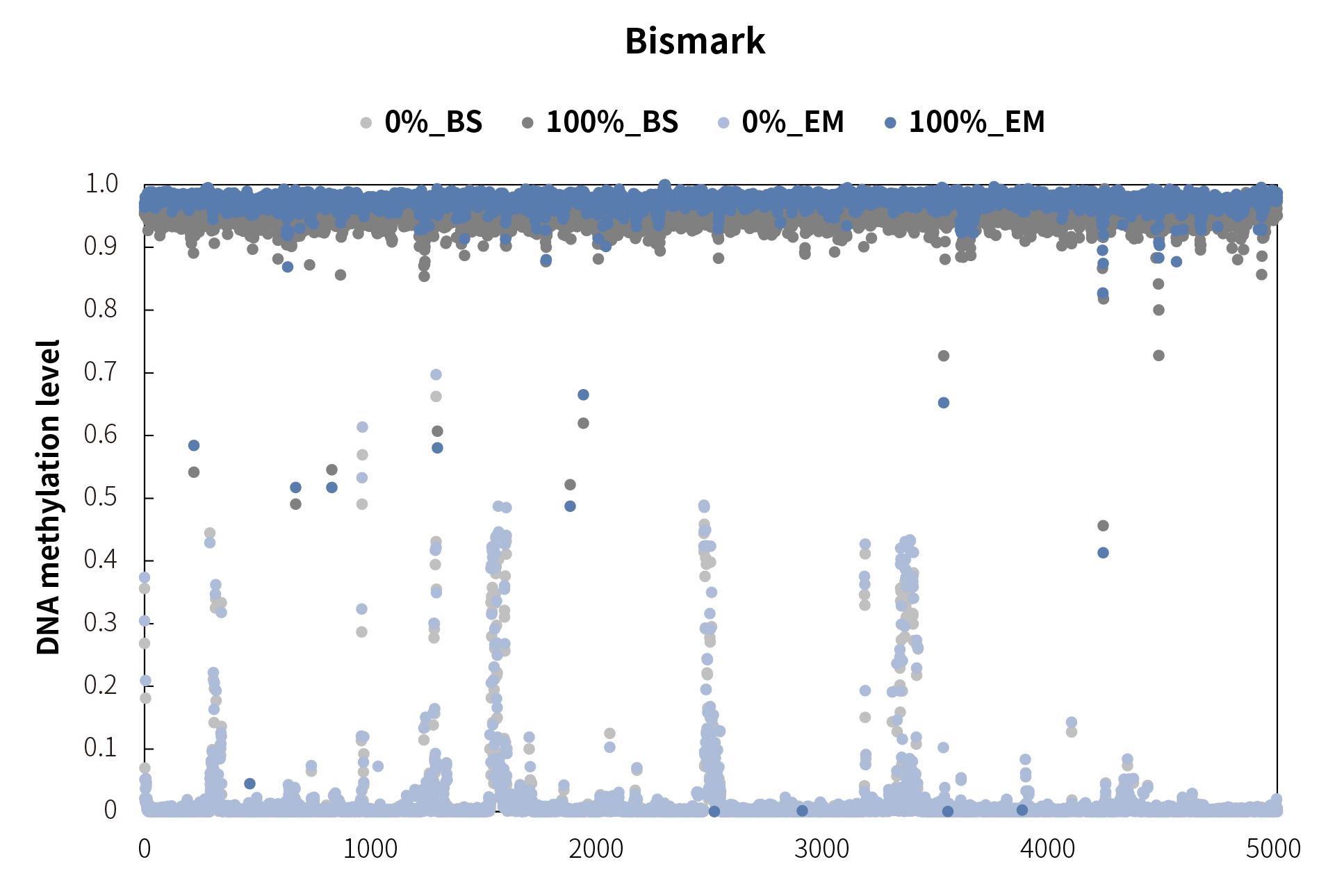
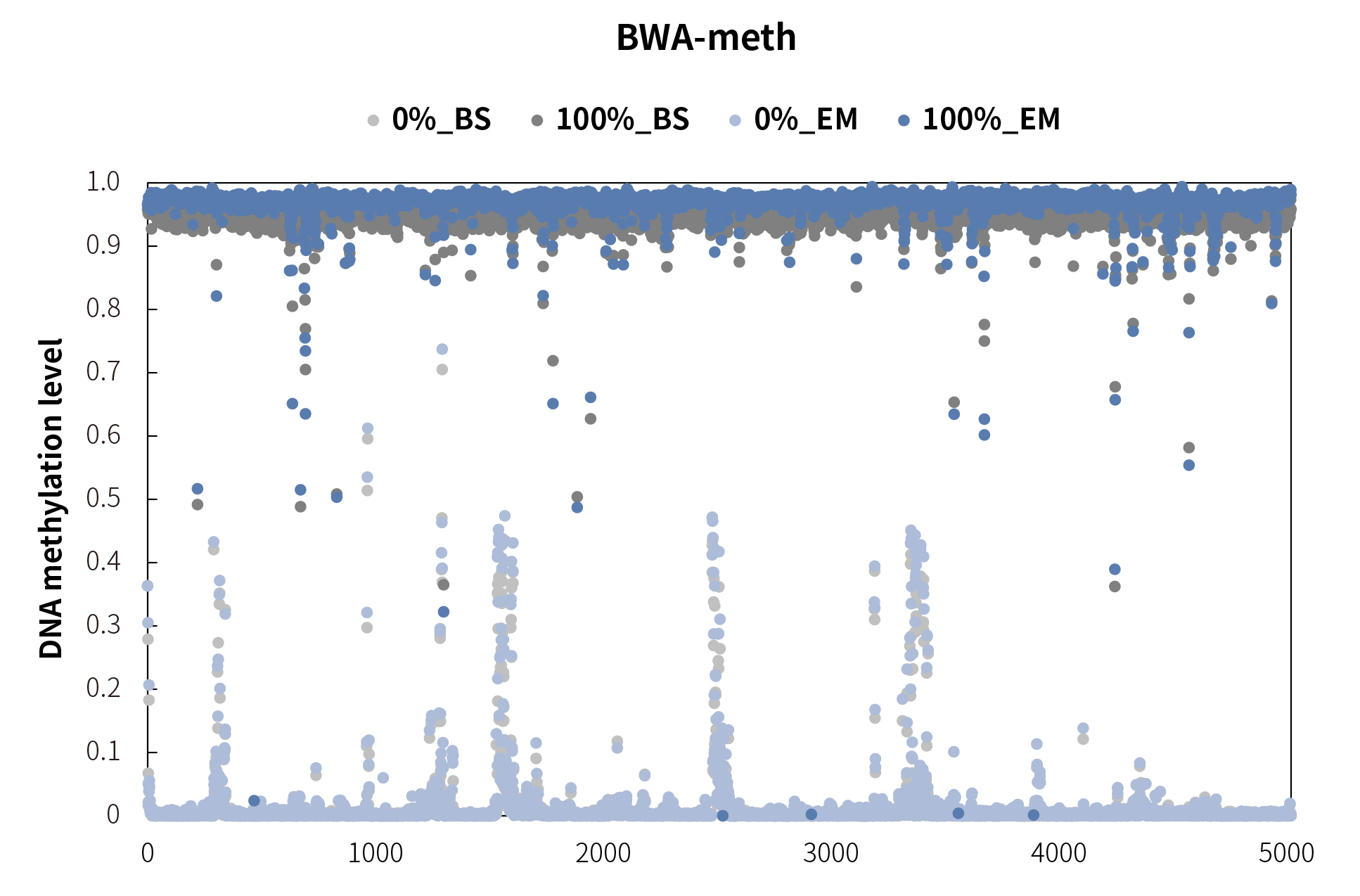
在这些 CpG 位点的甲基化程度散点图的结果中,BWA-meth 跟 Bismark 的 CpG 的甲基化检测结果基本吻合,而 Msuite 的甲基化程度的分布则有很大的区别,在多个位点均出现不一致情况。这很可能也是 Msuite 对甲基化位点基于深度统计的方式导致的,从而影响后续甲基化程度检测的灵敏性。
EM 转化方式中,0% 标准品 CpG 位点出现了不同程度的甲基化,我们进一步的分析显示,主要原因是这些位点为连续 CpG 区域且具有回文结构,由此影响了 EM 转化效率。
向下滑动查看
图 4. 标准品 CpG 位点不同软件检测甲基化程度结果
真实样本的甲基化检测
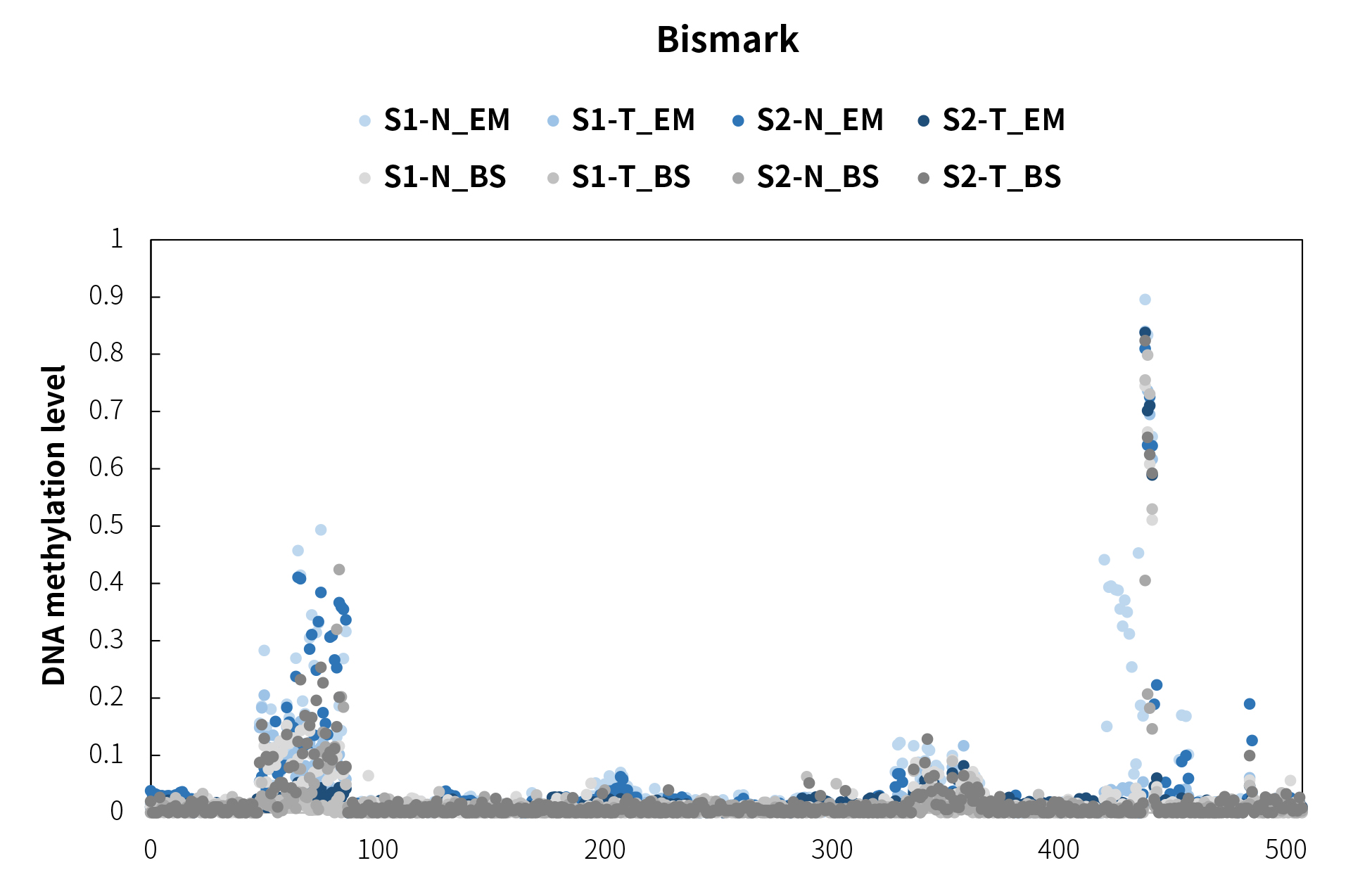
两例配对卵巢癌 FFPE 样本 S1 和 S2 使用 Meth-HR Panel 进行靶向甲基化测序分析,EM 转化的中靶率也显著高于 BS 转化(~70% vs ~50%)。不同比对软件分析 CpG 位点的甲基化程度散点图如图 5 所示,BWA-meth 和 Bismark 二者甲基化检测结果较为一致,而 Msuite 的甲基化程度的分布则有很大的区别。
向下滑动查看
图 5. 真实样本 CpG 位点不同软件检测甲基化程度结果
Bismark 的比对结果兼容性更优,我们先基于此软件的比对结果进行后续 DMR 分析。但结果显示三款 DMR 软件的分析一致性较差:eDMR 未检测到任何 DMR 信息;Methylkit 仅在 S1 样本 BS 转化的 PALB2 基因启动子区有检测到 DMR;DSS 软件则检测到 S1 样本 EM 转化的 BRCA1 启动子区的高甲基化(图6)和 S2-EM/S2-BS 的 HOX9 启动子区的高甲基化(图6)。其中,S1 样本后续经纳昂达 NanOnco Plus Panel v3.0(货号 1001111F)检测为 HR 通路相关基因阴性且被 HiSNP Ultra Panel v1.0 (货号 1001838) 检测判定为高 HRD 样本,这跟预期结果一致:BRCA1 启动子高甲基化导致的基因沉默。
DSS 软件搭配 Bismark 的结果,我们在 BWA-meth 比对结果下进行了重现。但是 S1 样本 BS 转化的结果始终未检测到 BRCA1 基因启动子区的高甲基化,这可能是由于 BS 转化 DNA 的高损害和较低的起始投入量影响了检测灵敏度,导致造成假阴性的结果,还有待于进一步验证。
表 3. 三款 DMR 软件分析结果
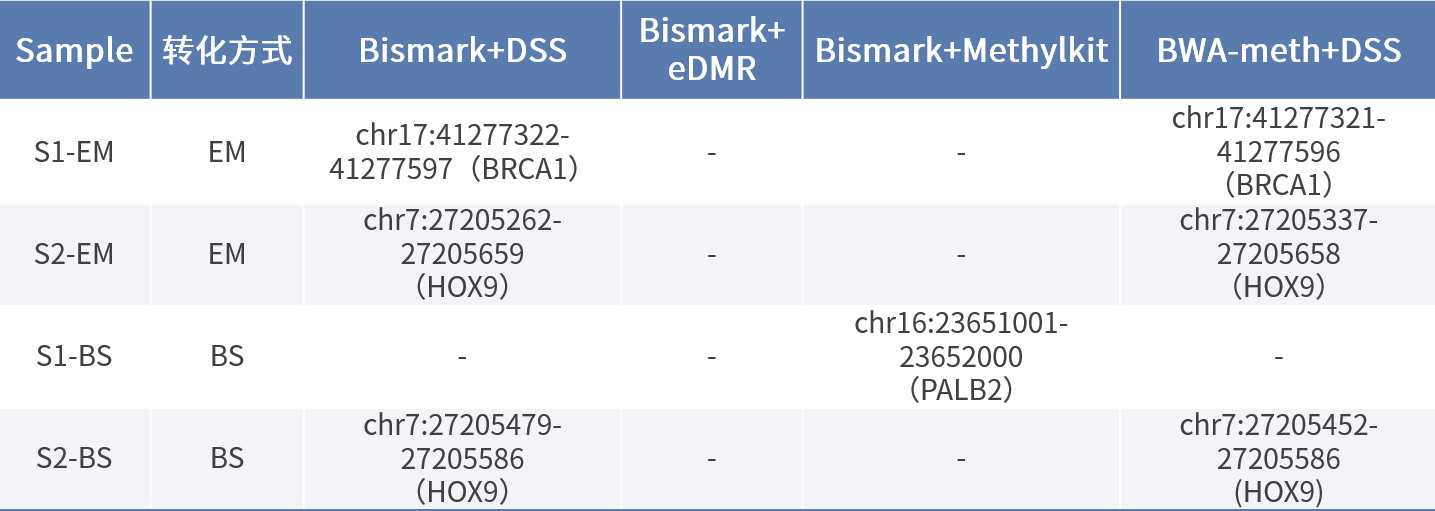


图 6. DMR 分析结果
总结
DNA 甲基化维持正常细胞功能、遗传印记、胚胎发育以及人类肿瘤发生中起着重要作用。靶向甲基化测序中的重亚硫酸盐法转化对样本损伤较大,使用酶法转化不仅可规避这一问题,应用表现也更优。酶转化法也适用于低起始量样本,从而保留更多有效的表观遗传学信息,尤其适用于肿瘤早筛中珍稀的 cfDNA。
基于纳昂达科技的靶向甲基化测序方案和示例,我们评估了不同的比对和 DMR 分析软件:BWA-meth 软件的比对率更高,对启动子中高 GC 区域有更好的兼容性;DSS 的 DMR 算法针对 BS 和 EM 转化有较高的检测灵敏度。后续我们也将继续抛砖引玉,展示更多甲基化单链建库和双链建库的评测结果,敬请期待!
进一步了解产品详情,请联系纳昂达当地销售或邮件发送至 sales@njnad.com。
纳昂达科技
纳昂达科技 成立于 2011 年,秉承 “Nano Trans More ”的核心理念和 “靶向精准,用心服务诊断”的奋斗宗旨,致力于为科研院校、医疗机构、临检单位、产业公司、测序服务商等提供专业化和高质量的靶向测序产品与闭环解决方案。
公司深耕精准靶向领域,目前拥有 MGI 和 Illumina 双测序平台多款文库构建试剂盒和全套液相杂交试剂产品。明星产品还包括全外显子 Panel、泛实体瘤和血液肿瘤 Panel 以及呼吸道病毒 Panel 等,并提供全面完善的双平台捕获探针定制化服务。
面积 > 2,000 平米的高通量测序研发中心和 > 2,500平米的GMP级别(YY/T0287-2017idt ISO13485:2016)体外诊断试剂生产基地为产品创新与生产质量保驾护航。纳昂达的销售网络覆盖全国并已外延至海外地区。
公司将与客户共成长,对客户的需求全力以赴,为全球用户提供靶向测序解决方案和 IVD 试剂原料。

Nanodigmbio
微信公众号 | nanodigmbiotech
参考文献
[1] Skvortsova K, Stirzaker C, Taberlay P. The DNA methylation landscape in cancer[J]. Essays in biochemistry, 2019, 63(6): 797-811.
[2] Vaisvila R, Ponnaluri V K C, Sun Z, et al. Enzymatic methyl sequencing detects DNA methylation at single-base resolution from picograms of DNA[J]. Genome research, 2021, 31(7): 1280-1289.
[3] Krueger F, Andrews S R. Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications[J]. bioinformatics, 2011, 27(11): 1571-1572.
[4] Pedersen B S, Eyring K, De S, et al. Fast and accurate alignment of long bisulfite-seq reads[J]. arXiv preprint arXiv:1401.1129, 2014.
[5] Sun K, Li L, Ma L, et al. Msuite: A High-Performance and Versatile DNA Methylation Data-Analysis Toolkit[J]. Patterns, 2020, 1(8): 100127.
[6] Akalin A, Kormaksson M, Li S, et al. methylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles[J]. Genome biology, 2012, 13(10): 1-9.
[7] Li S, Garrett-Bakelman F E, Akalin A, et al. An optimized algorithm for detecting and annotating regional differential methylation[C]. BMC bioinformatics. BioMed Central, 2013, 14(5): 1-9.
[8] Feng H, Conneely K N, Wu H. A Bayesian hierarchical model to detect differentially methylated loci from single nucleotide resolution sequencing data[J]. Nucleic acids research, 2014, 42(8): e69-e69.