PNAS | 泛着丝粒研究揭示大豆基因组中广泛的着丝粒重新定位
01 背景
着丝粒是真核生物染色体中的重要结构。着丝粒功能异常往往导致细胞在分裂过程中染色体无法正确分离,从而影响植物的生长和发育[1]。在大多数真核生物中,着丝粒是由组蛋白 H3 变体 CENH3 表观遗传学定义的[2],同时,着丝粒也是人工合成染色体的必需原料。因此,着丝粒结构和功能的解析不仅是染色体生物学领域亟待攻克的基础科学问题,也是未来合成生物学的必经之路。
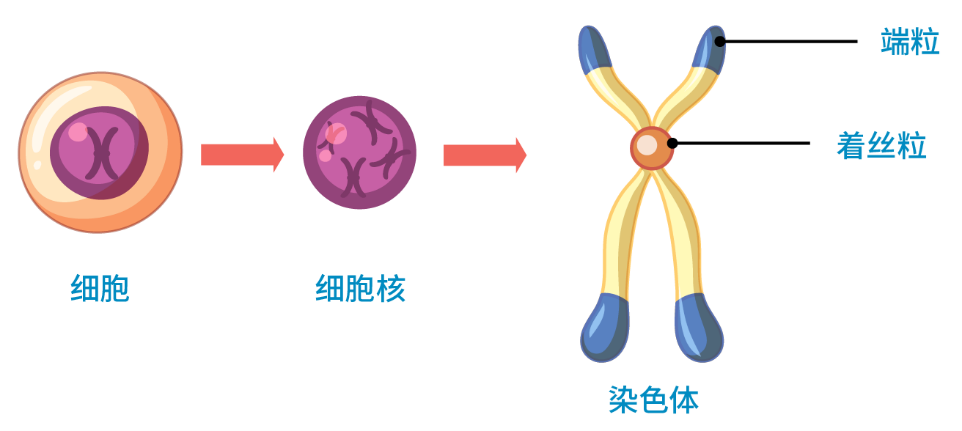
着丝粒存在高度的重复序列,且在序列组成和长度上存在物种差异,给其精确组装和功能解析带来了极大挑战。近年来,借助长读长测序技术及先进算法,研究揭示了人类和拟南芥着丝粒的进化模型。然而这些研究仅局限在个体水平,对群体水平上的着丝粒进化轨迹尚不清楚。着丝粒重新定位是一种不需序列重排即可形成全新着丝粒的现象,被用来解释新着丝粒 (evolutionary-new centromeres, ENCs) 的进化[3]。在进化过程中,ENCs 可以在染色体区域的异位位置出现。
大豆是全世界最重要的粮食和油料作物之一。现代栽培大豆大约在 5000 年前由其近亲野生大豆驯化,继而进一步改良而成。虽然野生大豆基因组测序促进了比较基因组和进化研究[4],然而群体内和群体间着丝粒结构和序列多样性尚未清晰了解。
2023 年 10 月 10 日,中国科学院遗传与发育生物学研究所韩方普研究组与田志喜研究组的研究成果以 “Pan-centromere reveals widespread centromere repositioning of soybean genomes” 为题发表于 PNAS (IF = 11.1)。研究团队通过比较基因组学探索了大豆着丝粒在遗传学 (序列组成) 和表观遗传 (CENH3 定位) 方面的多样性和动力学。尤其是通过对 27 种大豆的 CENH3-ChIP-Seq 分析,揭示了大豆基因组演化过程中新着丝粒的形成机制。其中测序方案的文库构建试剂全部来自纳昂达 (Nanodigmbio)。

02 研究方法
植物材料:27 个大豆品种分别种植 10 天后,采集幼苗叶片立即于液氮速冻,长期存储于 -80°C 下以进行后续分析。
染色质免疫沉淀测序 (ChIP-Seq):使用 NadPrep DNA Library Preparation Kit for Illumina (1002101, Nanodigmbio) 和 NadPrep UDI Adapter Kit Set C1 for Illumina (1003221, Nanodigmbio) 构建 ChIP 和 Input DNA 文库于 Illumina NovaSeq 进行测序。
数据分析:

03 结果
3.1 与 CENH3 核小体相关的卫星阵列的结构和表观遗传学
研究人员对包括 3 个野生品种、9 个地方品种和 15 个栽培品种在内的共 27 个大豆品种进行了着丝粒序列、结构及位置的分析。为了实现这一目标,研究中制备了能够识别大豆着丝粒特异组蛋白 CENH3 的抗体,并利用 ChIP-Seq 明确了大豆着丝粒的位置和大小。同时通过生物信息学分析结合荧光原位杂交技术 (FISH),研究人员鉴定了大豆着丝粒区域的特异重复序列。在之前对大豆中三个着丝粒卫星的发现基础上,研究人员发现了另外两个与 1 号染色体特异性关联的着丝粒卫星序列 (图 1.)。这些新的卫星重复序列揭示了 1 号染色体着丝粒结构在不同材料中的显著重排,从而影响到 CENH3 的定位。
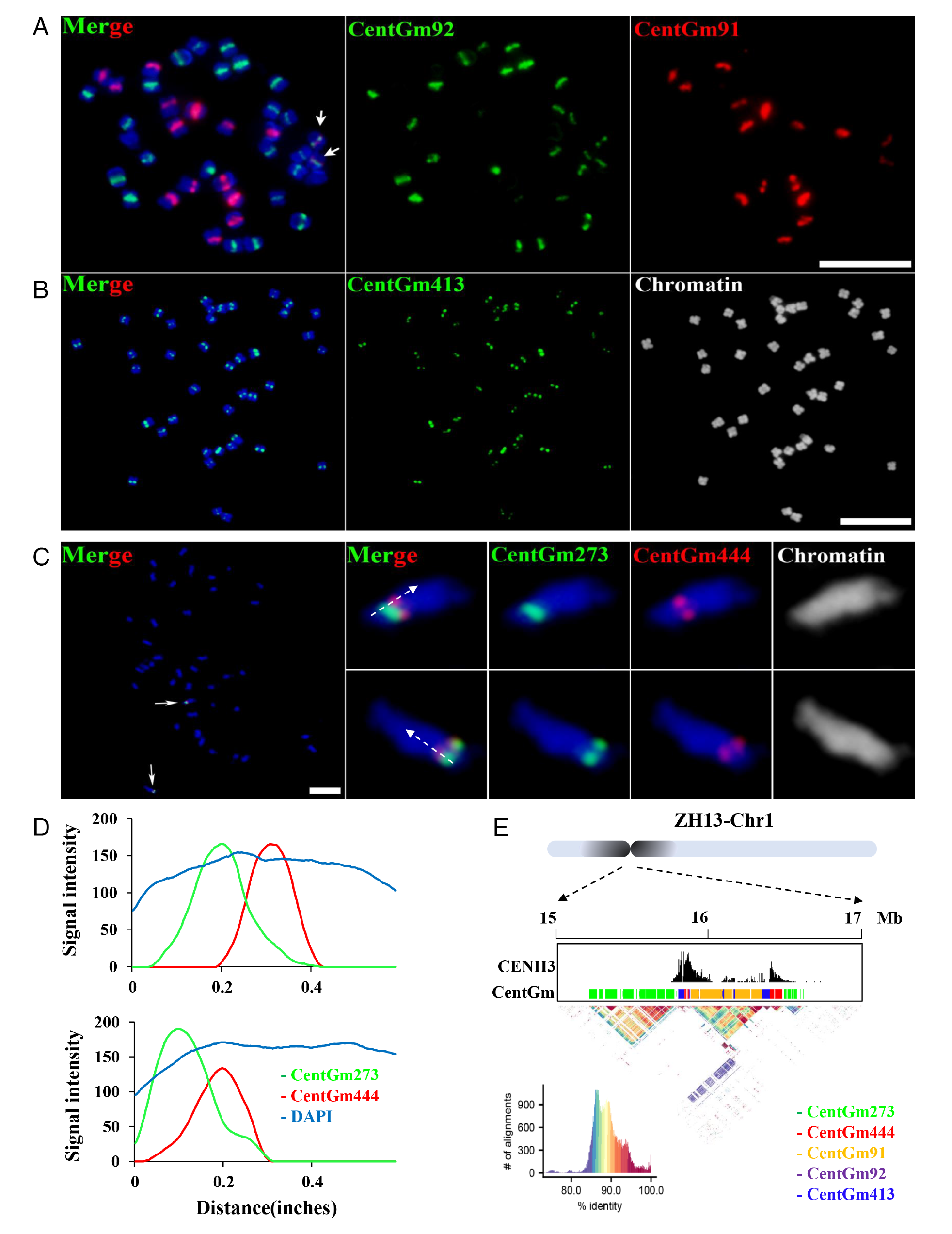
图 1. 在 ZH13 中与 CENH3 核小体相关的 DNA 的细胞学分析。A. ZH13 中 CentGm91 (红色) 和 CentGm92 (绿色) 的 FISH 信号 (标尺 10 μm);B. ZH13 中 CentGm413 (绿色) 的 FISH 信号 (标尺 10 μm);C. ZH13 中 CentGm444 (红色) 和 CentGm273 (绿色) 的 FISH 信号。方框内显示 1 号染色体的高倍放大图像。虚线表示用于量化信号强度的染色质部分 (标尺 10 μm)。D. 荧光图谱表明 CentGm273 和 CentGm444 不共定位。水平轴表示图 1.C 中虚线的起始位置 (单位:英寸),纵轴对应信号强度; E.在 ZH13 参考基因组的 Cen1 内注释 CentGm273 和 CentGm444 的不同位置不同图层分别展示了 CENH3 富集 [log2(ChIP/Input)],CenGm 注释以及配对卫星序列相似性。
3.2 大豆基因组进化过程中发生广泛的着丝粒重定位
为了研究所有 27 个品种在 20 条染色体上的着丝粒动态变化,研究人员将所有大豆品种的 CENH3-ChIP reads 与各自参考基因组比对,然后统一比对到 ZH13 参考基因组后,发现在 20 条染色体中,有 14 条染色体上发生了高频率的着丝粒重新定位 (图 2.)。着丝粒重新定位是指新的着丝粒形成在染色体的另一个位置,而没有发生 DNA 序列的重新排列。大多数新形成的着丝粒位于原有着丝粒位置附近,而一些则出现在与其亲缘关系较远的不同样本的相同位置出现。这表明它们的出现是相互独立的,而某些特定的染色体区域似乎更容易形成新的着丝粒。大豆及其他物种中广泛存在的着丝粒重定位现象暗示真正功能的着丝粒区域需要通过 CENH3-ChIP 实验确认,而不能单纯通过着丝粒特异序列的位置确定。此外,研究人员还进行了两个着丝粒位置不匹配的品种的杂交,以调查在杂交遗传背景中着丝粒位置会受到何种影响。研究人员发现大豆的着丝粒在 S9 代并没有重定位到全新的位置,它们仍然部分重叠在父母本的着丝粒区域上。此外,着丝粒区域的移动并非随机的,它们更倾向于结合在着丝粒重复序列上,这表明串联重复序列可能为 CENH3 提供了有利的环境,从而有助于维持着丝粒的稳定性。

图 2. 映射 C1、C3、L2 和 L3 的 CENH3 ChIP-seq 数据到它们各自的基因组。A-D. C1 (A)、C3 (B)、L2 (C) 和 L3 (D) 的 CENH3 ChIP-seq reads 比对到各自参考基因组。第一行显示了大豆着丝粒的 CENH3 富集 [log2(ChIP/Input)]。第二行显示了大豆着丝粒卫星的分布。第三行显示了转座元件 (TEs) 的分布。热图显示了在着丝粒上所有不重叠的 2 kb 区域中,大豆着丝粒卫星相对于其它卫星之间的序列同源性。
04 总结
着丝粒重定位现象,尤其是在植物中,一直以来都备受关注和调查。与动物相比,关于植物中着丝粒重定位的报告较少,但现有的研究为我们提供了有关这一过程的动态和机制的宝贵见解。尽管卫星在着丝粒功能中的作用机制尚不清楚,但显然各种因素,包括适当的转录水平、重组和非 B-形态结构,共同影响着丝粒的定位和稳定性。这都需要我们的科研人员进一步去探究,在探索着丝粒奥秘的这条路上,纳昂达一直与大家同行~
参考文献
[1]L. Comai, S. Maheshwari, M. P. A. Marimuthu, Plant centromeres. Curr. Opin. Plant Biol. 36, 158–167 (2017).
[2]J. Zhou et al., Centromeres: From chromosome biology to biotechnology applications and synthetic genomes in plants. Plant Biotechnol. J. 20, 2051–2063 (2022).
[3]Schubert, What is behind “centromere repositioning”? Chromosoma 127, 229–234 (2018).
[4]F. M. Piras et al., Uncoupling of satellite DNA and centromeric function in the Genus Equus. PLOS Genet. 6, e1000845 (2010).







