来吧,Panel 起来! - XCapert 探针设计平台
背景
靶向捕获测序在对基因组部分区域富集后,再进行核苷酸变异、插入缺失、拷贝数变异和基因重排等检测,可有效降低测序成本。靶向富集主要实现方式有两种,一是通过多重 PCR 直接扩增靶区域,二是通过寡核苷酸探针,对预文库杂交捕获实现。由于多重 PCR 存在交叉扩增和扩增效率不均一等原因,适用场景有限。基于探针 (Probe & Panel) 的杂交富集方式,既兼容多种变异类型检测,也易于优化,获得较佳的测序均一性,因而得到了广泛应用[1]。
针对通用需求,已有多家公司提供目录化 Panel 及杂交捕获方案,例如全外显子和泛癌种应用。直接购买商品化 Panel 比从头设计更加值得推荐,因为这些 Panel 通常已经过验证和优化。但当某些实验或者检测需要针对特定基因、固定疾病或者特殊信号通路时,则需要定制 Panel 的设计合成。定制 Panel 设计需要考虑到靶区域的选择、探针位置和方式,这些因素既与捕获效果直接相关,也直接影响合成的成本 (表 1)。
表 1. Panel 设计时纳入考量的因素

XCapert 在线设计平台
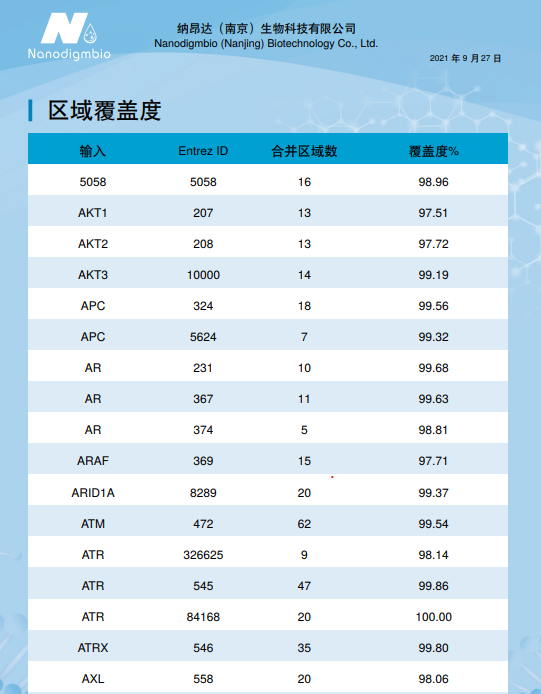
为了提供更加高效的 Panel 设计服务,纳昂达基于 > 100 万条探针验证优化的算法,近期推出了 XCapert 在线设计平台 (http://nadprobe.njnad.com/),可提供针对人源参考基因组的 DNA 捕获和 RNA 捕获的 Panel 设计流程,同时也包括甲基化和微生物方向的应用设计。用户不仅可自动化获取专业化设计结果,结合 NGS-QC based 质检和配套杂交试剂,也更容易、快速展开靶向捕获实验。
图 1丨XCapert 在线探针设计云平台
轻松输入
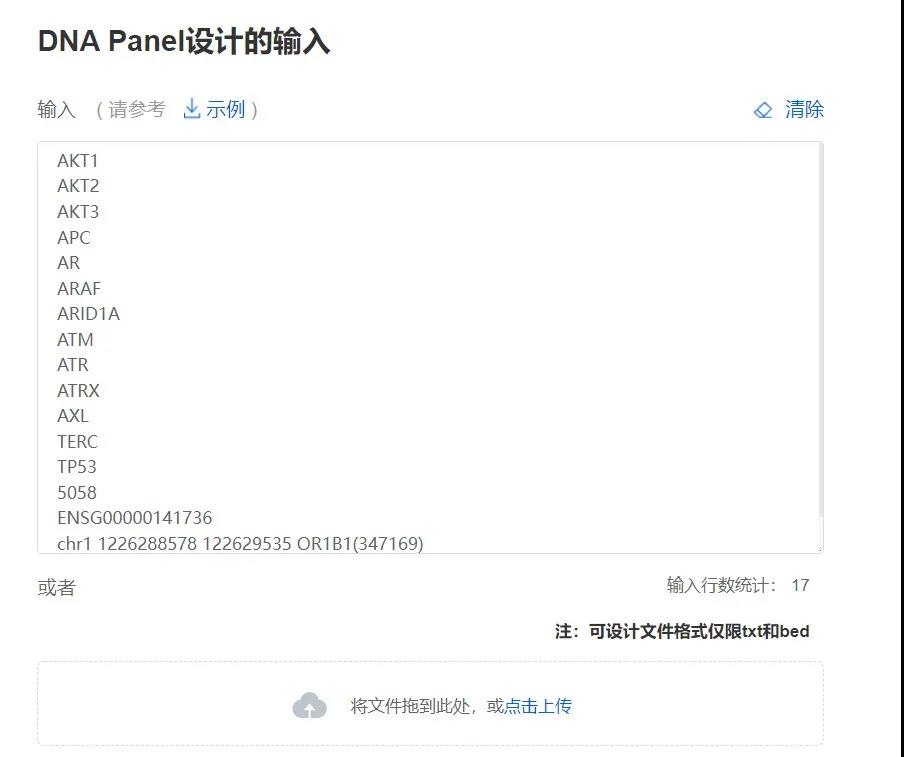
XCapert 兼容多种数据库格式的输入,例如基因名、基因 ID、Ensembl ID、SNP 位点、转录本 NM 号、CCDS 以及 Bed 格式,并可实现智能识别。用户直接将靶区域信息粘贴至文本框或以 TXT 文件上传,即可完成输入。如输入区域不正确或存在基因同名的情形,平台则识别后供选择确认,而无需反复调整输入 (图 2)。
左右滑动查看
图 2丨探针设计内容输入格式示例
如果靶区域含有人基因组上的重复序列,如Tandem repeats 或 Interspersed repeats,设计出的探针因特异性不佳,将导致中靶率急剧降低。XCapert 平台采用 homology-based 的方法,在对各数据库收录的重复序列基础上评估序列相似性,实现对靶标区域的分级屏蔽。一种是 remove all risk genome region,指去除靶区域中所有风险区域,从而保证探针的特异性并提升捕获中靶率,但可能降低对部分靶区域的覆盖度;另一种是 remove high risk genome region,指仅去除高风险区域,此时设计结果将包含安全探针和风险探针,需要用户适度进行探针分管,便于优化和改进。这样做的好处是可最大限度提升覆盖程度,又兼顾捕获中靶率 (图 3)。
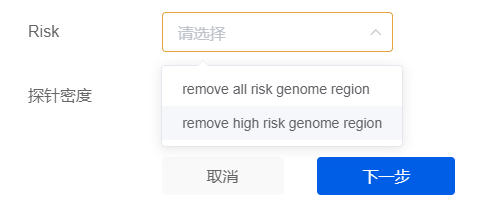
图 3 | 常见发生融合的 ROS1 intron 区域分别选择 remove high risk 和 remove all risk 时,2 种重复序列屏蔽模式结果存在显著区别。
DNA & RNA Panel 设计
DNA 和 RNA Panel 应用场景不同,分别靶向基因组和转录本,故设计思路和算法也不同。当选取 2x 设计时,DNA Panel (2x) 以靶区域上游 60 bp 为起点,每隔 60 bp 放置一条探针;RNA Panel (2x) 先将给定基因的全部转录本按 60 bp 间隔进行 2x 设计,然后再通过算法进行合并、去重和再设计,实现探针去冗余,从而以最少的探针数达到最佳的覆盖效果 (图 4)。
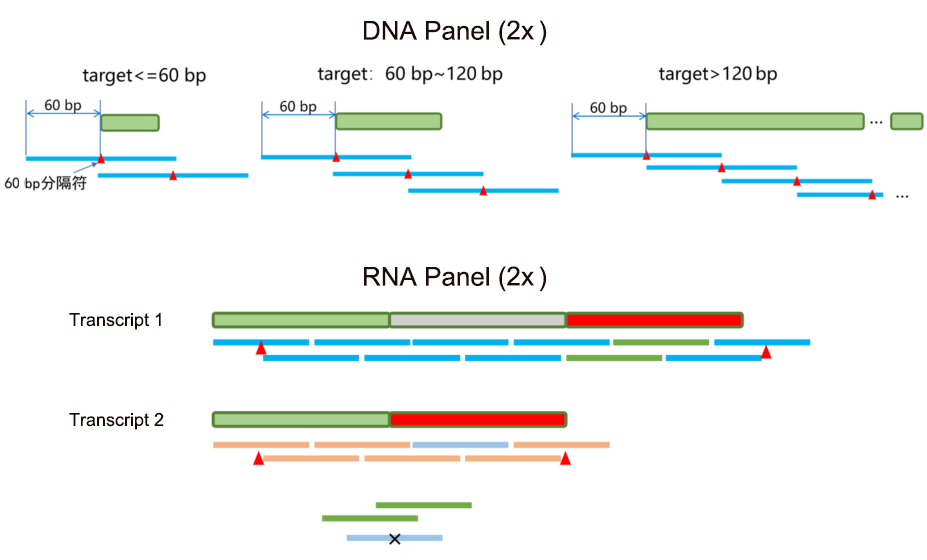
图 4 | DNA Panel 和 RNA Panel 设计示例
MSI Panel 设计
微卫星序列多为短串联重复序列,常规设计通常难以达到理想的捕获结果。以微卫星位点 BAT-40 为例,当使用常规的叠瓦式设计(图 5,Normal Design),捕获结果在重复序列区域存在明显降低,而优化方案则可大幅提升捕获效率,实现更好的均一性(图 5,Optimized Design)。XCapert平台针对特殊变异类型,例如 MSI、HLA、长片段插入缺失和基因重排等区域,可进行自动检测和优化设计[2],从而让这些难点区域捕获更加精准,让变异无处躲藏。
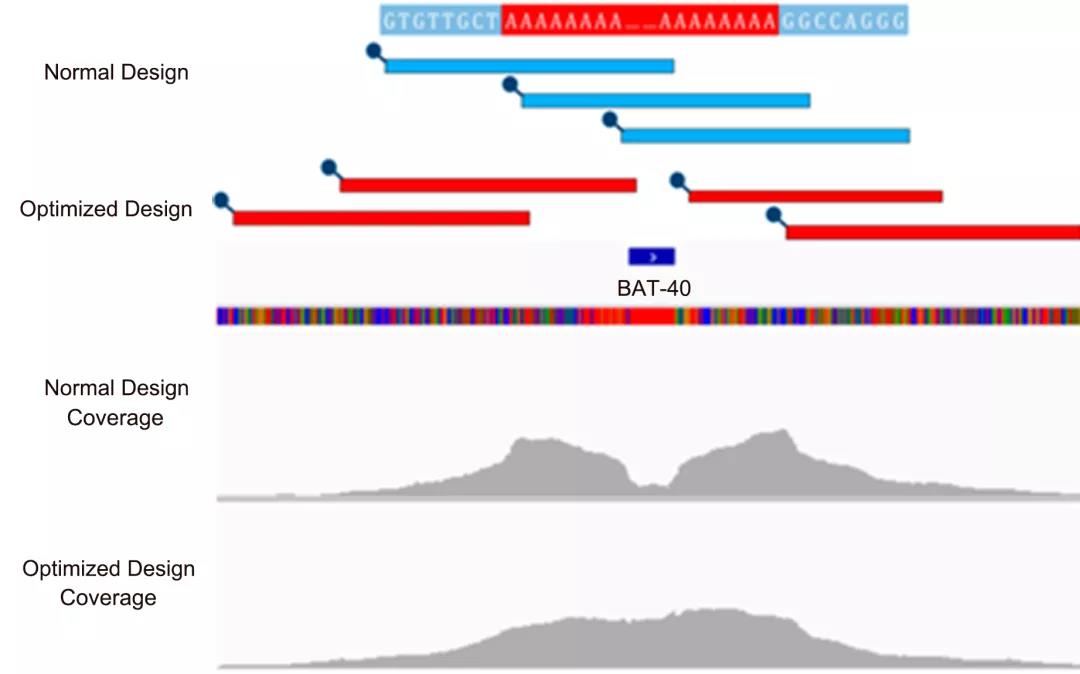
图 5 | 微卫星位点 BAT-40 设计示例
快速获取
目前 XCapert 探针在线设计平台(http://nadprobe.njnad.com) 已开放,且面向所有人免费,简单注册后即可使用 DNA 探针、RNA 探针和甲基化应用。常规设计下,10 min 内即可得到高质量的探针设计方案和报告 (图6)。
左右滑动查看
图 6 | 探针设计结果示例
平台内还有设计补充、下单维护等功能,设计完成后即可实现一键下单,也便于日后查看更新。
参考文献
[1] Kozarewa I, Armisen J, Gardner A F, et al. Overview of target enrichment strategies[J]. Current protocols in molecular biology, 2015, 112(1): 7.21. 1-7.21. 23.
[2] Dapprich J, Ferriola D, Mackiewicz K, et al. The next generation of target capture technologies-large DNA fragment enrichment and sequencing determines regional genomic variation of high complexity[J]. BMC genomics, 2016, 17(1): 1-14.