双低样本的成功建库是如何炼成的?单链建库技术助力成功率 up up up!
01 背景
文库制备是所有 NGS 工作流程中至关重要的一环,其本质是将待测 DNA/RNA 样本通过机械破碎、酶切等方法处理成合适长度,然后在片段两端连接特定的测序接头,从而形成符合测序平台要求的有效文库。dsDNA 文库制备常见的接头连接方式包括:T-A 连接,转座酶,分步连接和平末端连接。然而,dsDNA 文库制备方法用于较短片段 DNA、降解 DNA、ssDNA 以及带有碱基损伤、切刻、缺口的 DNA 样本时,往往效果不佳[1]。
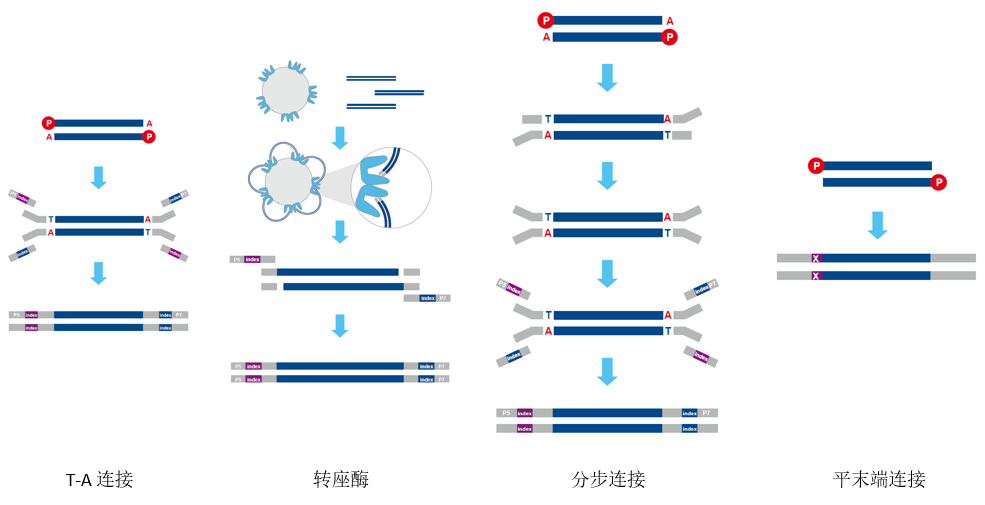
图 1. 常见的 dsDNA 建库接头连接方式。
ssDNA 文库制备方法已被证明能够有效保留更多较短、降解和碎片化的 DNA 信息[2],被认为是处理低质量、微量或降解较为严重的样本进行文库构建的可靠路径。其建库策略在于,首先将样本中的 DNA 高温变性为 ssDNA,然后在 ssDNA 的两端分步添加接头并形成 dsDNA 结构,之后通过 PCR 扩增,实现文库的构建。
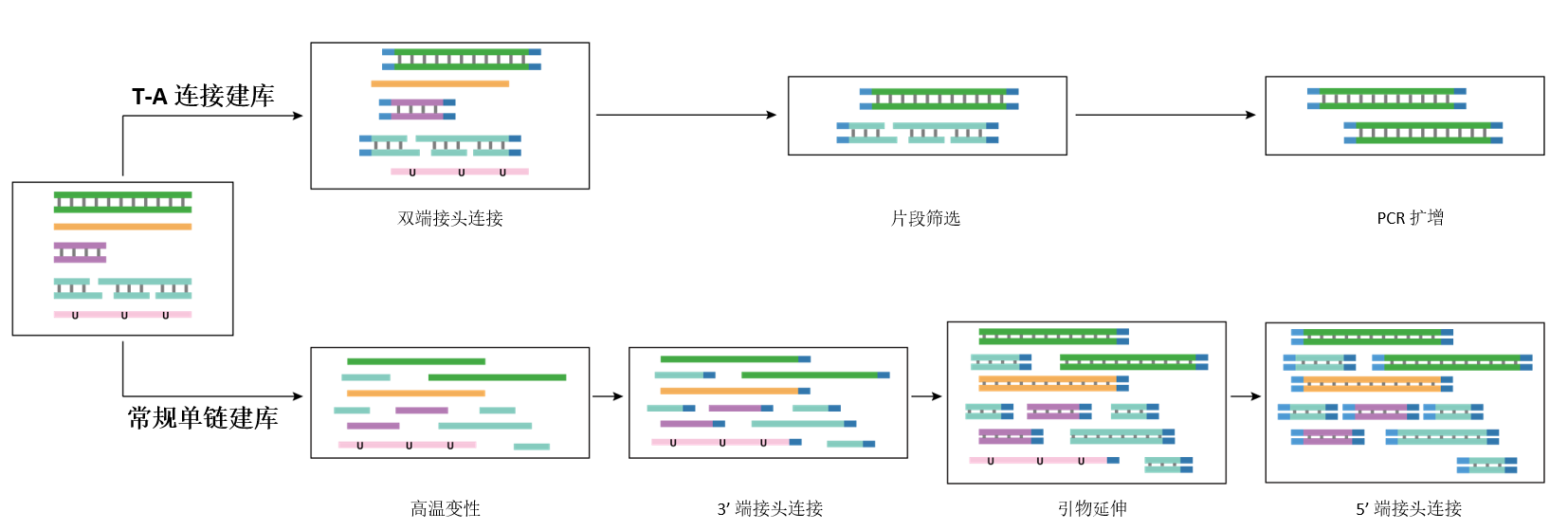
图 2. T-A 连接建库 vs. 常规 ssDNA 建库对不同 DNA 样本的处理能力。
单链建库技术摆脱了传统建库基于双链 T/A 连接的依赖,因此不再受限于 DNA 末端完整性、双链完整性、DNA 起始量以及接头/样本浓度比例等多种因素,从而实现了 DNA 连接效率以及 DNA 模板利用率的最大化,特别是在处理古 DNA、cfDNA/ctDNA、FFPE DNA、ChIP DNA 以及亚硫酸氢盐转化产物等样本时具有显著的富集优势[3-5],从而提高了古 DNA 研究、基于液体活检的癌症基因组/遗传学等研究的成功机会。
02 ssDNA 通用型文库制备解决方案 (for Illumina®)
2.1方案简介
ssDNA 通用型文库制备方案 (for Illumina®) 基于高效的单链连接原理,专为 Illumina® 高通量测序平台而开发。该方案特别适用于低质量样本和微量样本的文库构建,起始量可从 10 pg 到 250 ng,适用于全基因组测序、全基因组甲基化测序,同时兼容液相杂交靶向捕获测序。经过优化设计,该方案可兼容 cfDNA、gDNA、FFPE DNA,及其亚硫酸氢盐转化后的产物。
2.2方案流程
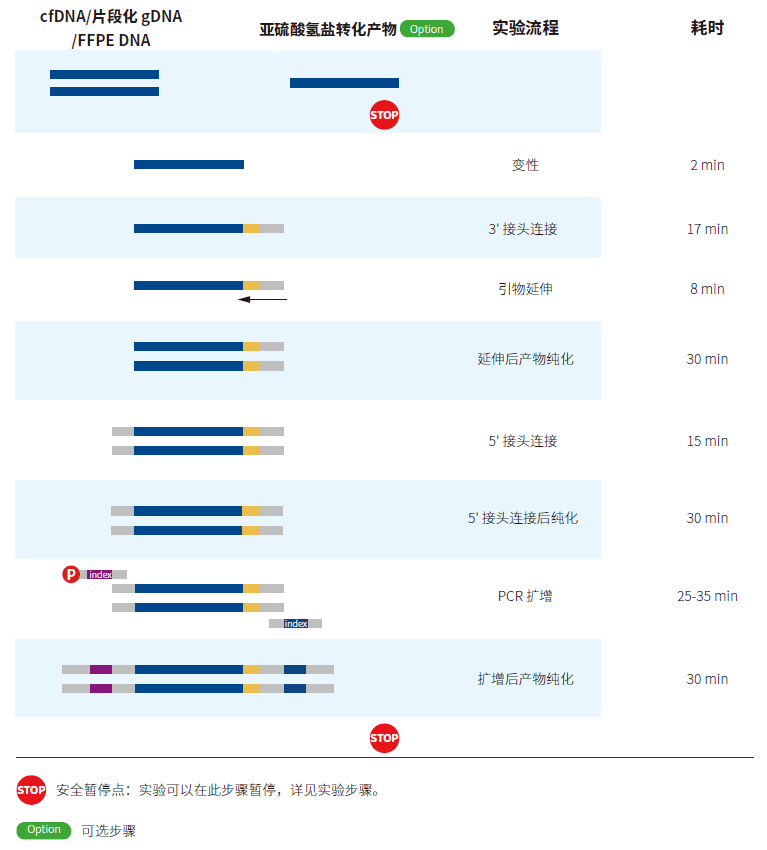
图 3. ssDNA 通用型文库制备方案流程。
2.3方案特点
宽泛的样本起始量:适应宽泛的样本起始量范围 (10 pg-250 ng),相较传统双链建库技术更具优势。
兼容多元化样本类型:广泛兼容各种样本类型,包括 gDNA、cfDNA、FFPE DNA 及其亚硫酸氢盐转化产物,普适性强。
全面兼容多等级 FFPE 样本:支持高度降解的低质量 FFPE,变“废”为“宝”。
低起始量/微量样本高效建库:支持低起始量/微量 gDNA 和 cfDNA/ctDNA 等样本,文库产出稳定高效。
高精度甲基化建库解决方案:为亚硫酸氢盐转化产物提供建库方案,满足甲基化研究的高精度数据需求。
03方案表现
3.1兼容不同起始量样本
ssDNA 通用型文库制备方案 (for Illumina®) 支持投入量 10 pg-250 ng 的稳定建库。使用人类基因组 DNA 标准品 (Promega,G1521)
(图 4. A) 及其亚硫酸氢盐转化产物 (图 4. B) 分别进行测试,投入量由 10 pg 到 250 ng 不等,参照推荐 PCR 循环数进行扩增,两种类型的 gDNA 在各投入量下均能得到较为理想的文库产量。
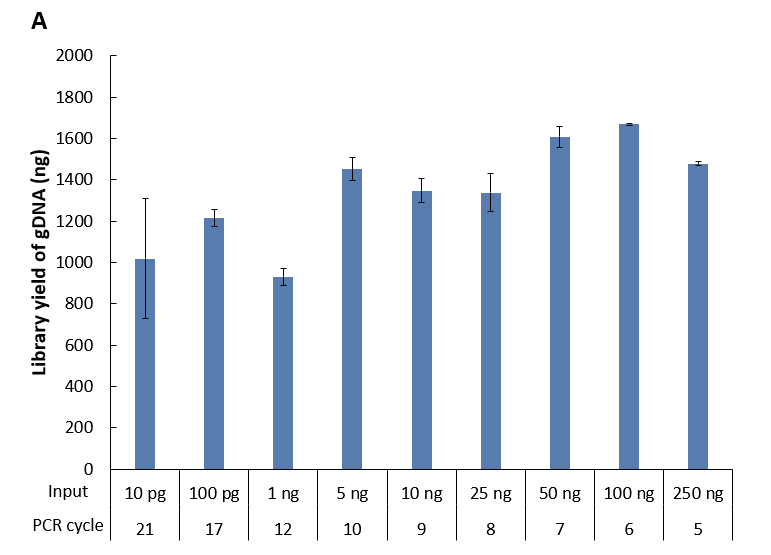
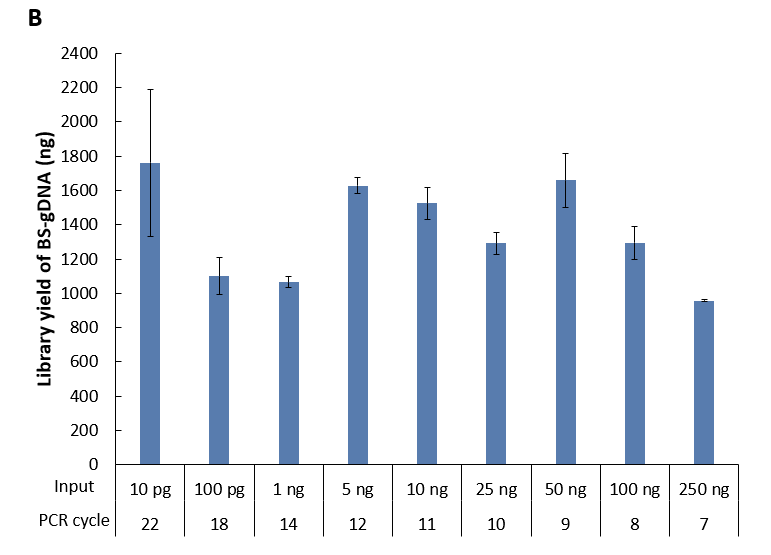
图 4. ssDNA 通用型文库制备方案对不同样本在不同起始量下的建库产出。A. gDNA 样本;B. gDNA 亚硫酸氢盐转化产物。
3.2兼容多类型样本
gDNA、FFPE DNA 和 cfDNA 及其亚硫酸氢盐转化产物分别使用 ssDNA 通用型文库制备方案 (for Illumina®) 进行单链文库构建,均能得到较为理想的文库产量 (图 5. A),且文库片段分布较为集中,从片段分布中没有观察到明显的接头二聚体产生 (图 5. B)。
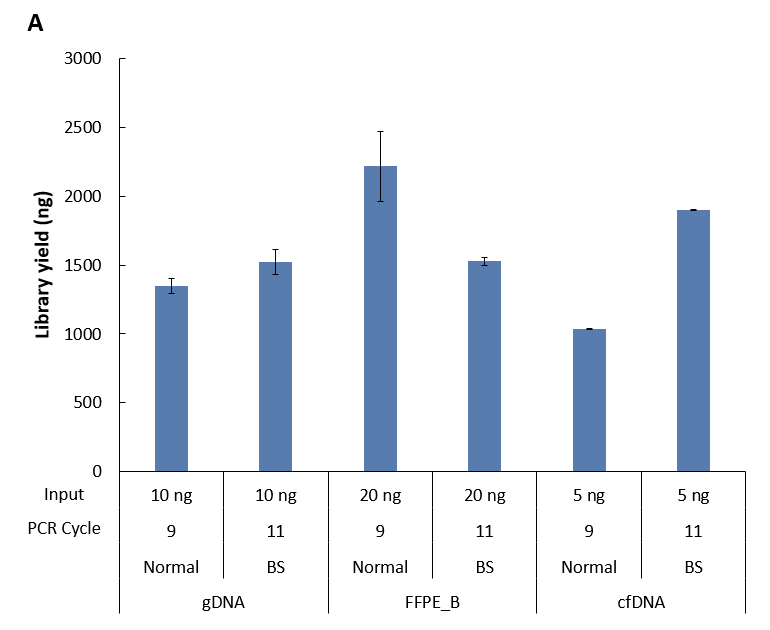
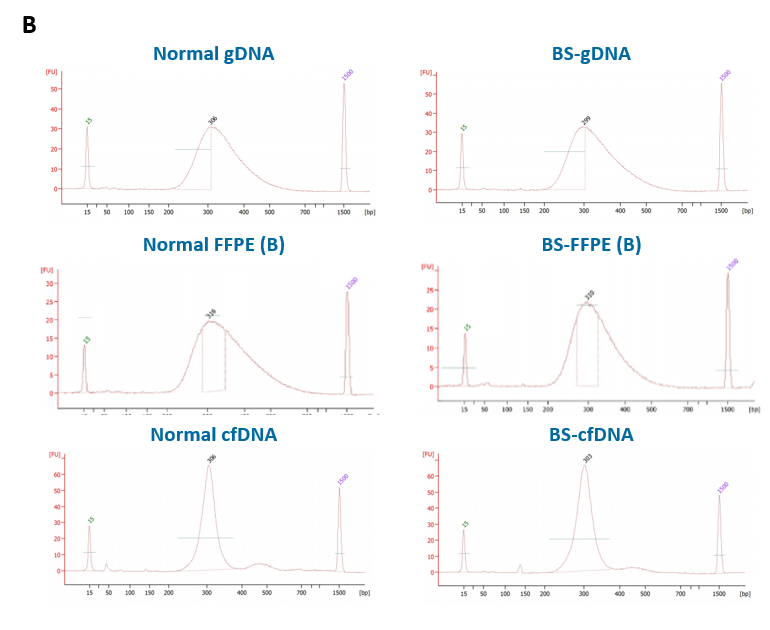
图 5. ssDNA 通用型文库制备方案对不同类型样本的建库产出。A. 文库产量;B. 文库片段分布。
注:FFPE 样本为 B 级 FFPE DNA。分级标准 | FFPE_B,~15 kb 有隐约可见主带,同时存在中度弥散。
3.3超低等级 FFPE 样本高效建库
使用 ssDNA 通用型文库制备方案 (for Illumina®) 对 B、C、D 等级的 FFPE 样本进行单链建库,并同时与双链建库方式进行对比。结果显示,在同一起始投入量下,随着 FFPE 样本质量等级的降低,其文库产量有所下降;但是,针对超低等级的 FFPE 样本 (FFPE_D) 在较低起始投入量的情况,单链建库较双链建库具有更高的文库产量,表现出明显的优势 (图 6. A)。除此之外,单链建库靶向测序的优势随着 FFPE 样本质量等级的降低愈发显著。具体而言,单链建库在比对率、中靶率较双链建库更高 (图 6. B),对靶区域的覆盖度也更优 (图 6. C),去重后的平均测序深度也会更高 (图 6. D),尤其对 FFPE_D 样本的去重后测序深度达到双链建库的 4 倍。
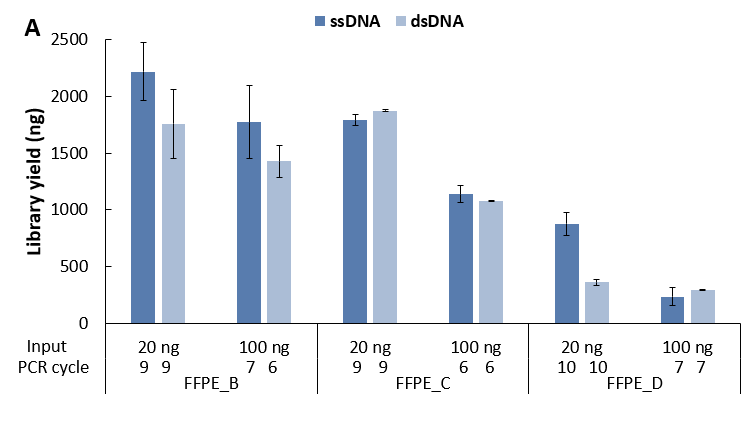
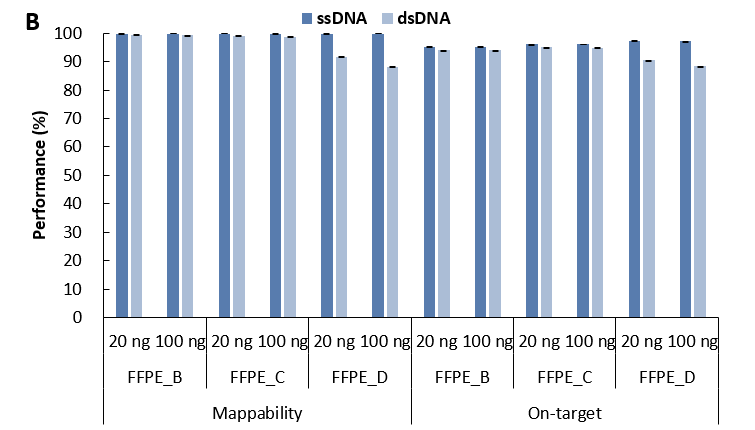
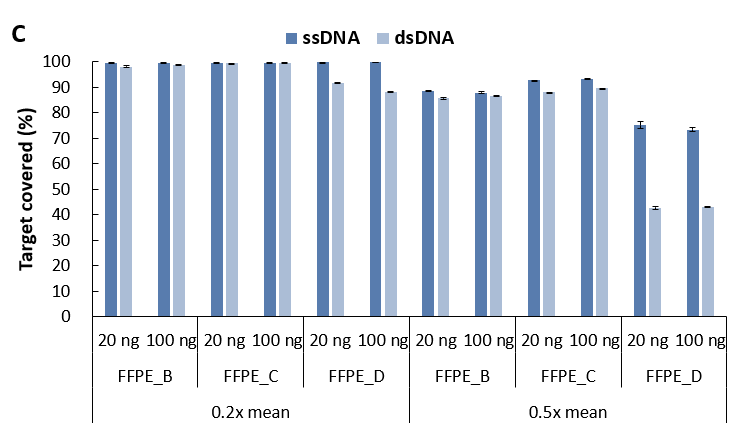
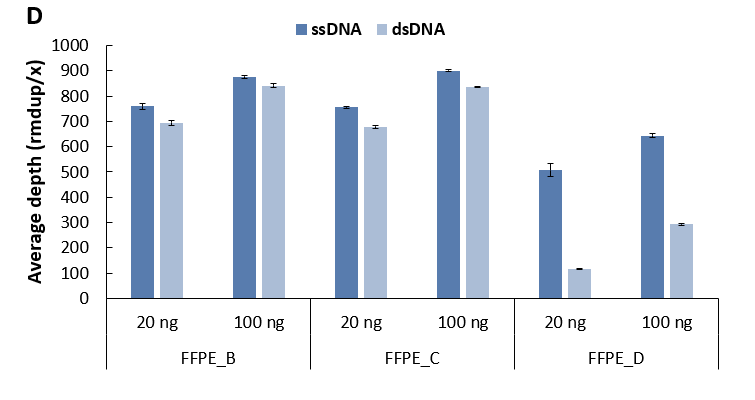
图 6. 不同文库制备方案对不同等级 FFPE DNA 样本的建库产出和捕获表现比较。A. 文库产量;B. 比对率 & 中靶率; C.靶区域覆盖度; D.平均测序深度 (去重后)。以 NadPrep® Hybrid Capture Reagents 和 LungCancer Panel v1.0 进行杂交捕获。
注:FFPE DNA 分级标准 |
FFPE_B,~15 kb 有隐约可见主带,同时存在中度弥散;FFPE_C,在 200-2500 bp 之间存在弥散状分布,无清晰主带;FFPE_D,主要分布在 250-1000 bp,呈弥散状。
3.4低起始量样本高效建库
3.4.1 gDNA
与传统双链建库相比,单链建库在样本低起始量投入时表现出显著优势。对 gDNA 标准品分别进行单双链建库,从文库产量来看 (图 7. A),在投入量 0.1
ng 时,单链建库表现出绝对优势,其文库产量高出双链建库数十倍,且靶区域覆盖度也显著更优 (图 7. C),单链建库在各投入量下的去重后平均测序深度均高于双链建库 (图 7. D),这有助于我们对样本信息进行更准确的捕获,从侧面节省了测序数据量,从而降低了检测成本。
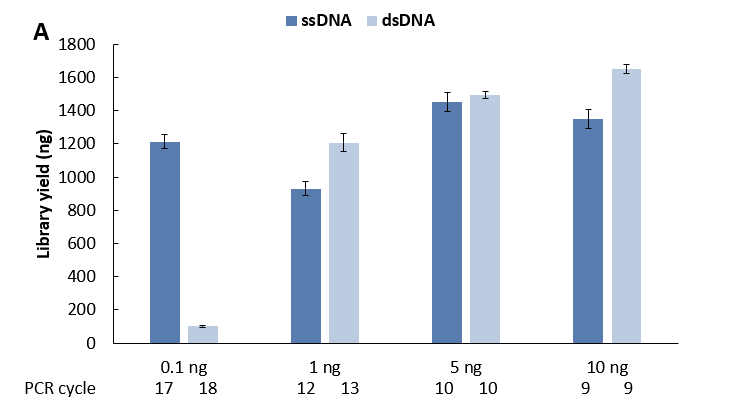
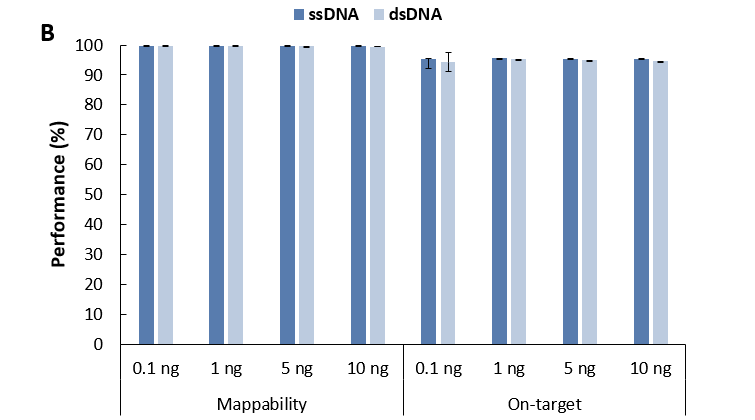
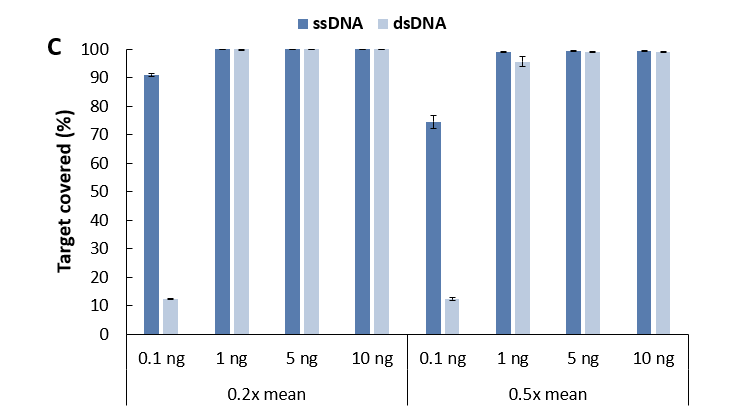
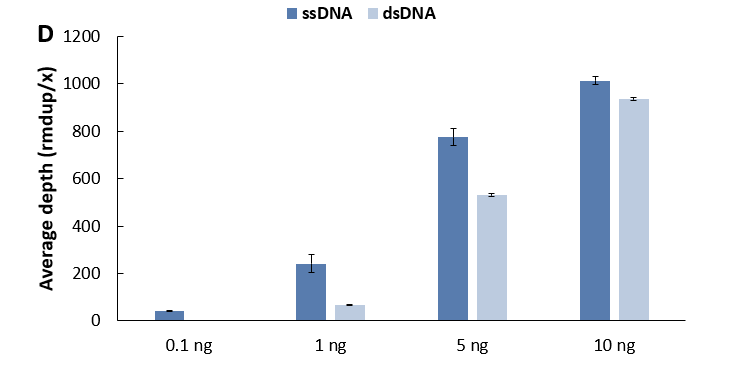
图 7. 不同文库制备方案对微量 gDNA 样本的建库产出和捕获表现比较。A. 文库产量; B.比对率 & 中靶率; C. 靶区域覆盖度; D. 平均测序深度 (去重后)。以 NadPrep® Hybrid Capture Reagents 和LungCancer
Panel v1.0 进行杂交捕获,0.78 Gb 数据量进行分析。
3.4.2 cfDNA
同样,我们对 cfDNA 低投入量时的建库情况也进行了测试,结果与 gDNA 保持一致 (图 8.)。对于小于 10 ng 的低起始量样本建库时,高效连接的单链建库弥补了双链建库的短板,是一种更合适的建库方式选择。
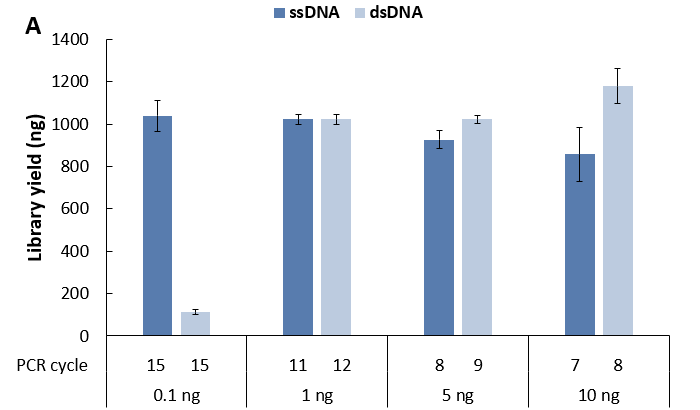
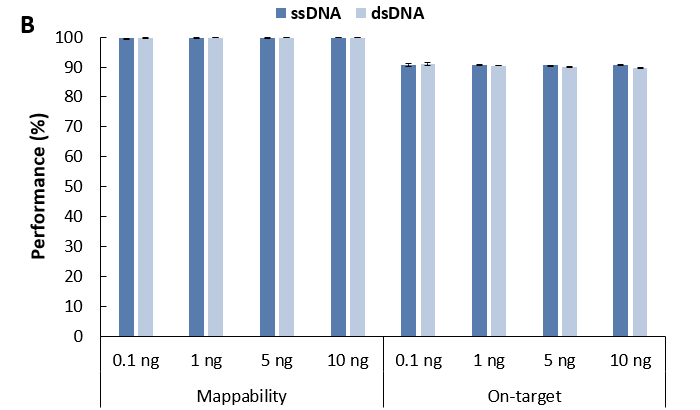
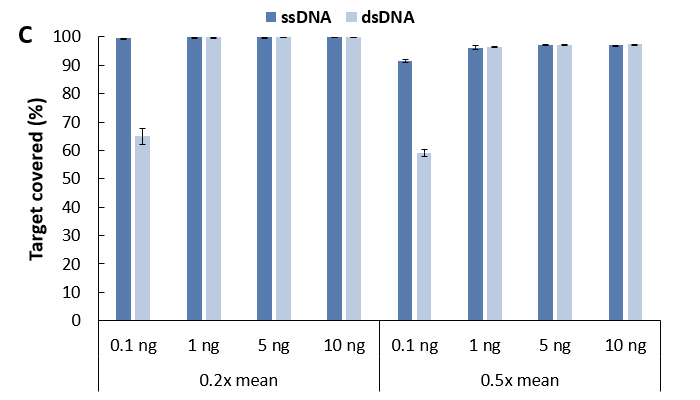
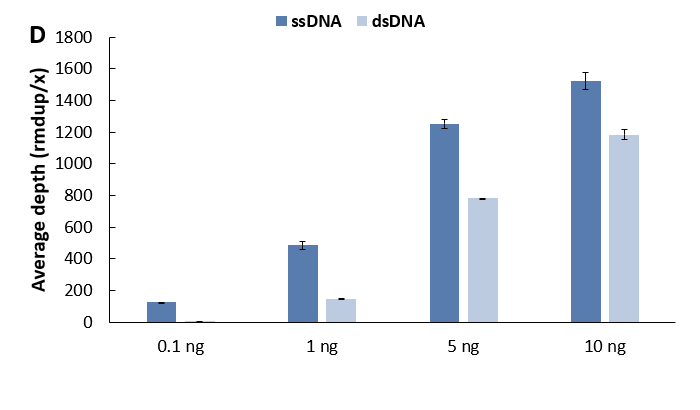
图 8. 不同文库制备方案对微量 gDNA 样本的建库产出和捕获表现比较。 A. 文库产量; B. 比对率 & 中靶率; C. 靶区域覆盖度;D. 平均测序深度 (去重后)。以 NadPrep® Hybrid Capture Reagents 和LungCancer
Panel v1.0 进行杂交捕获,0.81 Gb 数据量进行分析。
ssDNA 通用型文库制备方案 (for Illumina®) 除了在低投入量及低质量样本中具有显著优势外,在甲基化建库方面更是毫不逊色,从而为肿瘤早筛提供新的方案。更多测试数据请关注纳昂达,我们将在下期中为大家展示更多单链建库的魅力。
参考文献
[1] Gansauge M T, Meyer M. Single-stranded
DNA library preparation for the sequencing of ancient or damaged DNA[J]. Nature
protocols, 2013, 8(4): 737-748.
[2] Zhu J, Huang J, Zhang P, et al.
Advantages of single-stranded DNA over double-stranded DNA library preparation
for capturing cell-free tumor DNA in plasma[J]. Molecular diagnosis &
therapy, 2020, 24: 95-101.
[3] Burnham P, Kim M S, Agbor-Enoh S, et
al. Single-stranded DNA library preparation uncovers the origin and diversity
of ultrashort cell-free DNA in plasma[J]. Scientific reports, 2016, 6(1):
27859.
[4] Stiller M, Sucker A, Griewank K, et al.
Single-strand DNA library preparation improves sequencing of formalin-fixed and
paraffin-embedded (FFPE) cancer DNA[J]. Oncotarget, 2016, 7(37): 59115.
[5] Mouliere F, Robert B, Arnau Peyrotte E,
et al. High fragmentation characterizes tumour-derived circulating DNA[J]. PloS
one, 2011, 6(9): e23418.







